REVISÃO INTEGRATIVA
CONCEIÇÃO, Maria Gabriella [1], FRANÇA, Camilla Estêvão De [2], CONCEIÇÃO, Sandra Maria Da Penha [3]
CONCEIÇÃO, Maria Gabriella. FRANÇA, Camilla Estêvão De. CONCEIÇÃO, Sandra Maria Da Penha. Identificação de microRNAs modificados em doenças autoimunes. Revista Científica Multidisciplinar Núcleo do Conhecimento. Ano 05, Ed. 11, Vol. 09, pp. 114-120. Novembro de 2020. ISSN: 2448-0959, Link de acesso: https://www.nucleodoconhecimento.com.br/veterinaria/doencas-autoimunes, DOI: 10.32749/nucleodoconhecimento.com.br/veterinaria/doencas-autoimunes
RESUMO
Descobertos há pouco mais de uma década em Caenorhabditis elegans, os miRNAs (classe de genes de RNAs não codificantes de 22 nucleotídeos) são hoje reconhecidos como reguladores fundamentais da expressão gênica em plantas e animais. Na literatura, os transcritos de RNA não codificantes superam o grupo de sequências de codificação de proteínas e prometem novas descobertas e explicações para fenômenos biológicos essenciais e patologias. Recentemente, estudos estabelecem uma conexão de miRNAS a imunossupressão por possuírem um significativo papel na regulação do sistema imunológico. Em vista disso, este trabalho teve por objetivo correlacionar e identificar através de uma revisão integrativa miRNAs alterados em doenças autoimunes, na tentativa de entender seu mecanismo durante o desenvolvimento dessas doenças, para contribuir na inovação de métodos e elaboração de estratégias visando o tratamento dessas doenças, bem como, reconhecer miRNAs biomarcadores que possam ser utilizados para o diagnóstico e monitoramento do tratamento de doenças autoimunes. A partir de artigos analisados, foi possível concluir que existem alterações e relações específicas entre os microRNAs e doenças autoimunes. Comprovando que os miRNAs apresentam um papel fundamental na regulação do sistema imune podendo ser aplicados no processo de imunossupressão. Deve se realizar a quantificação de cada expressão de miRNAs correlacionada em cada doença autoimune para obtenção de um resultado apurado para estabelecer miRNAs como biomarcadores.
Palavras-Chaves: MicroRNAs, doenças autoimunes, biomarcadores, imunossupressão.
1. INTRODUÇÃO
Os mecanismos epigenéticos de controle de expressão genica são fenômenos que, por impor um padrão de expressão genica específico e herdado pela progênie sem alterar a sequência de nucleotídeos no DNA, seriam capazes de promover a especialização de células e suas linhagens (PUGH et al., 2012). Os principais mecanismos epigenéticos incluem a mutilação de DNA, modificação de histonas e RNAs regulatórios não codificantes, os quais interagem entre si para regular a conformação estrutural da cromatina (SUZUKI e BIRD, 2012).
A classe de genes de RNAs não codificantes de 22 nucleotídeos é denominada como microRNAs (miRNAs). Os miRNAs regulam a expressão de genes alvo ligando-se a locais complementares em transcritos mais longos codificados em genomas de animais, plantas e vírus (BARTEL, 2004; MOLNAR et al., 2007; ZHAO et al., 2007).
Na literatura, os transcritos de RNA não codificantes superam o grupo de sequências de codificação de proteínas e prometem novas descobertas e explicações para fenômenos biológicos essenciais e patologias (HA, 2014). Recentemente, estudos correlacionam miRNAS a imunossupressão por possuírem um significativo papel na regulação do sistema imunológico. Pois atuam na direção e a distinção das células do sistema imune inato e adaptativo, monitorando a manutenção e a evolução de progenitores imunes bem como a diferenciação e a função de células imunes maduras efetoras (HARTEL et al., 2004).
Uma vez que o epi genoma tem um papel crítico na determinação do programa de expressão gênica (HOWE et al., 2013), diferenças na expressão gênica entre os indivíduos pode afetar a resposta a fármacos, e pode ser causada por diferenças no epi genoma (SZYF, 2004).
Sabendo-se que indivíduos possuem respostas diferentes aos imunossupressores (HARTEL et al., 2004) e que alterações epigenéticas podem estar relacionadas com a regulação dessas respostas, a proposta desse estudo visa caracterizar a expressão de microRNAs em doenças autoimunes podendo permitir entender as consequências intracelulares da imunossupressão nas células do sistema imune e fornecer uma nova metodologia para avaliar a ativação do sistema imune em pacientes. Além disso, a pesquisa contribui para a identificação de marcadores de expressão diferencial de miRNA, que permitam a priori distinguir indivíduos respondedores dos não respondedores ao tratamento com fármacos imunossupressores.
2. DESENVOLVIMENTO
Para o desenvolvimento do objetivo proposto, foi realizada uma revisão integrativa na base acadêmica do Google (https: //scholar.google.com.br) e na base de armazenamento de artigos Scientific Eletronic Library (Scielo). Houve também uma consulta na plataforma PubMed (https://www.ncbi.nlm.nih.gov/pubmed) para a obtenção de bibliografia.
A pesquisa foi baseada em estudos de autores que abordavam as palavras-chave, microRNA e Lupus; e Vitiligo; e Psoriase; e Crohn´s; e doença de Graves e entre outros estudos pertinentes ao assunto.
No processo de seleção, foram escolhidos artigos escritos em inglês, português ou espanhol. Posteriormente os artigos mais recentes compostos pelos principais critérios e palavras-chave, foram avaliados e contabilizados, possibilitando a construção de uma tabela que exemplifica o número de artigos encontrados e a quantidade de microRNAs mostrados que apresentaram um nível de expressão (baixa, alta e normal) em doenças autoimunes.
Tabela 1– Caracterização da quantidade de artigos identificados em bases cientificas que correlacionam microRNAs e doenças autoimunes de acordo com o nível de expressão.
| PALAVRAS CHAVE | Nº DE ARTIGOS | UP | DOWN | ALTERADOS |
| MicroRNA and Lupus | 10 | 7 | 2 | 11 |
| MicroRNA and Vitiligo | 8 | 5 | 6 | 5 |
| MicroRNA and Psoríase | 17 | 9 | 2 | 3 |
| MicroRNA and Grave´s | 10 | 6 | 4 | 2 |
| MicroRNA and Crohn´s | 5 | 5 | 0 | 1 |
| Total | 50 |
Fonte: Conceição (2018)
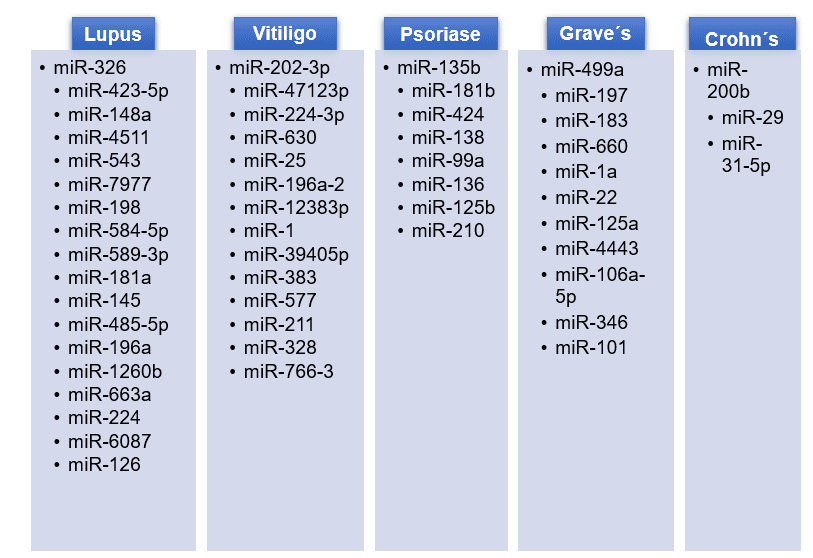
Como resultados, foi possível identificar e criar uma tabela com os 68 tipos específicos de miRNAs analisados na literatura que se apresentaram alterados e modificados nas doenças autoimunes de Lupus, Vitiligo, Psoriase, Grave´s e Crohn´s.
Tabela 2- Identificação de microRNAs específicos expressados em cada doença autoimune.
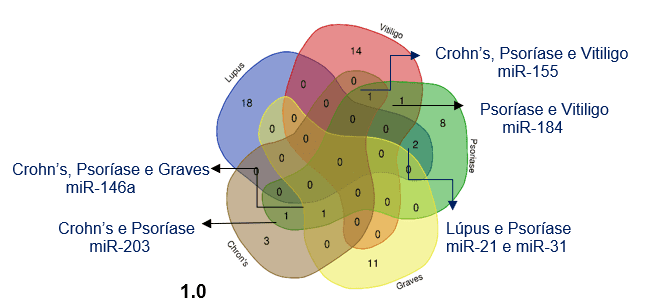
Partindo dos resultados apresentados na tabela, foi utilizada a ferramenta de diagrama de venn para a caracterização do estabelecimento da relação de cada miRNA expressado em todas as enfermidades citadas. Nele, é possível verificar que o miR-155 aparece com um alto nível de expressão nas doenças Crohn´s, Psoríase e Vitiligo. Foi verificado também que nas doenças de Lúpus e psoríase o miR-21 e miR-31 obtiveram um valor significativo de expressão. Já o miR- 184 apresenta um vínculo entre Psoríase e Vitiligo. Em Crohn’s e Psoríase constatou-se o miR-203 como conexão. E por fim, o miR-146a foi relacionado as doenças de Crohn’s, Psoríase e Graves.
Figura 1 – Diagrama de Venn associando microRNAs específicos em determinadas doenças autoimunes.
Com os resultados formulados e caracterizados acima, esta pesquisa serve como auxílio para diversos estudos envolvendo mecanismos epigenéticos e gera a possibilidade da verificação e quantificação apurada da expressão de cada microRNA descrito para o desenvolvimento de biomarcadores em cada enfermidade citada como potenciais alvos para o tratamento dessas doenças.
3. CONSIDERAÇÕES FINAIS
Alterações na expressão de miRNAs foram relacionadas com doenças autoimunes sugerindo que os miRNAs tem um papel importante na regulação do sistema imune podendo ser aplicados no processo de imunossupressão. Em análise geral observa-se a existência de poucas produções cientifica com essa temática. Esta revisão serve de auxílio para os profissionais e estudantes da área da saúde no contexto de desenvolvimento de trabalhos científicos voltados para este tema.
REFERÊNCIAS
BARTEL, David P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell, v. 116, n. 2, p. 281-297, 2004.
HA, Minju; KIM, V. Narry. Regulation of microRNA biogenesis. Nature reviews Molecular cell biology, v. 15, n. 8, p. 509-524, 2014.
HARTEL, Christoph et al. Sensitivity of whole-blood T lymphocytes in individual patients to tacrolimus (FK 506): impact of interleukin-2 mRNA expression as surrogate measure of immunosuppressive effect. Clinical chemistry, v. 50, n. 1, p. 141-151, 2004.
HOWE, Kerstin et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature, v. 496, n. 7446, p. 498-503, 2013.
MOLNÁR, Attila et al. miRNAs control gene expression in the single-cell alga Chlamydomonas reinhardtii. Nature, v. 447, n. 7148, p. 1126-1129, 2007.
PUGH, Trevor J. et al. Medulloblastoma exome sequencing uncovers subtype-specific somatic mutations. Nature, v. 488, n. 7409, p. 106-110, 2012.
RODRIGUEZ, Antony et al. Identification of mammalian microRNA host genes and transcription units. Genome research, v. 14, n. 10a, p. 1902-1910, 2004.
SUZUKI, Miho M.; BIRD, Adrian. DNA methylation landscapes: provocative insights from epigenomics. Nature Reviews Genetics, v. 9, n. 6, p. 465-476, 2008.
SZYF, Moshe. Toward a discipline of pharmacoepigenomics. Current Pharmacogenomics, v. 2, n. 4, p. 357-377, 2004.
ZHAO, Tao et al. A complex system of small RNAs in the unicellular green alga Chlamydomonas reinhardtii. Genes & development, v. 21, n. 10, p. 1190-1203, 2007.
[1] Graduação em Medicina Veterinária.
[2] Doutorado em Morfologia. Mestrado em Ciência
[3] Orientadora. Doutorado em andamento em Ciências da Saúde. Mestrado em Ciências da Saúde. Especialização em andamento em Enfermagem em Saúde do Trabalhador. Especialização em MBA em Serviços de Saúde-Área Hospitalar. Especialização em Educação Profissional na Área da saúde. Especialização em Enfermagem em Obstetrícia. Aperfeiçoamento em Aprimoramento para Enfermeiros da Rede Básica de S. Graduação em Enfermagem.
Enviado: Julho, 2020.
Aprovado: Novembro, 2020.
