ARTÍCULO ORIGINAL
MELO, Nataly Sayonara da Silva [1], SILVA, Maria Goretti Varejão da [2], ALMEIDA, Anna Carolina Soares [3], MEDEIROS, Anna Karolyne de Araujo [4], SILVA, Daniel Dias da [5], SOUZA, Paula Mariana Salgueiro de [6], SILVA, Marcela Oliveira da [7], SOARES, Anísio Francisco [8], MENDONÇA, Marcelo [9], MEDEIROS, Elizabeth Sampaio de [10]
MELO, Nataly Sayonara da Silva, et al. Salmonella spp. Multifármaco virulento y resistente recuperado de canales de pollo en Brasil. Revista Científica Multidisciplinar Núcleo do Conhecimento. Año. 08, Ed. 04, Vol. 01, págs. 92-114. Abril 2023. ISSN: 2448-0959, Enlace de acceso: https://www.nucleodoconhecimento.com.br/biologia-es/multifarmaco-virulento, DOI: 10.32749/nucleodoconhecimento.com.br/biologia-es/multifarmaco-virulento
RESUMEN
El objetivo de este estudio fue evaluar la producción de biofilm, el perfil de susceptibilidad y la detección de genes de resistencia presentes en aislados de Salmonella spp de canales frescas de pollo vendidas en una metrópoli brasileña. De un total de 61 muestras de canales frescas de aves de corral, 21 fueron positivas para la presencia de Salmonella spp. En cuanto a la prueba de sensibilidad antimicrobiana, (13/21) los aislamientos probados fueron resistentes a al menos un antibiótico, correspondiente al 61,9%, y el 38% (08/21) fueron resistentes a múltiples fármacos. Se identificaron al menos dos genes de resistencia en todos los aislamientos, especialmente los genes relacionados con la resistencia a β-lactamasas y quinolonas. También se observó que algunos aislados de Salmonella spp mostraron patrones genéticos idénticos. Y los 21 aislados fueron capaces de formar biopelícula. La identificación del biofilm de Salmonella spp. formando y portando diferentes genes de β-lactamasa y determinantes de resistencia a quinolonas demuestra la capacidad de estas bacterias para acumular diversos mecanismos de virulencia y resistencia a antimicrobianos. Por lo tanto, la propagación de diferentes grupos clonales de Salmonella spp. MDR en canales de carne de aves de corral expresada en este atestiguan la necesidad de controles efectivos para contener este microorganismo, que además de ser un riesgo para la salud pública, también es responsable de considerables pérdidas económicas.
Palabra clave: Betalactámicos, Intoxicación alimentaria, PCR, Mercado público.
INTRODUCCIÓN
La salmonelosis es una enfermedad transmitida por los alimentos causada por cepas patógenas de Salmonella spp. Los casos de salmonelosis en humanos ocurren principalmente a través del consumo de alimentos o agua contaminados. En la mayoría de los casos, los alimentos de origen animal, especialmente los de aves de corral, son las principales fuentes de infecciones por Salmonella spp. Los síntomas de la infección entérica son náuseas, vómitos, diarrea no sanguinolenta, fiebre, resfriado, dolor abdominal, mialgia y dolores de cabeza, y en pacientes inmunodeprimidos puede provocar bacteriemia, endocarditis y muerte (GRIMONT y WEILL, 2007).
Según los Centros para el Control y la Prevención de Enfermedades (CDC), 1,2 millones de casos de infecciones, 23.000 hospitalizaciones y 450 muertes por año en los Estados Unidos de América son causados por Salmonella spp, aproximadamente. Y las infecciones relacionadas con la ingestión de alimentos contaminados representan aproximadamente 1.0 millones de casos (CDC, 2020). En Brasil, según el Ministerio de Salud, se notificaron un total de 12.660 casos de enfermedades transmitidas por los alimentos entre 2000 y 2017, y Salmonella spp. fue uno de los principales agentes notificados en estos casos, lo que representa un total del 35% de los casos notificados (BRASIL, 2019).
En la mayoría de los casos de salmonelosis, los individuos sanos no necesitan tratamiento. Sin embargo, los pacientes inmunodeprimidos necesitan pasar por tratamiento (SERENO et al., 2017). Sin embargo, el uso indiscriminado de antimicrobianos, especialmente en la producción animal, ha llevado a un mayor número de microorganismos resistentes a los agentes terapéuticos, lo que lleva a un número limitado de opciones antimicrobianas, lo que puede resultar en fracasos del tratamiento (ALEKSHUN y LEVY, 2007; PENG et al., 2014). A lo largo de los años, ha habido un aumento en la prevalencia de cepas de Salmonella resistentes a múltiples medicamentos, principalmente en productos avícolas, como la carne de ave (AZEVEDO, 2014; BAPTISTA et al., 2018; DUARTE et al., 2009; FITCH et al., 2016; BONI et al., 2011; REZENDE et al., 2005; RIBEIRO et al., 2007; THRELFALL, 2002).
Por lo tanto, la aparición de Salmonella multirresistente (MDR) es una preocupación mundial, debido al aumento de las tasas de hospitalización y muerte. Este fenómeno es consecuencia del uso extensivo de antibióticos por parte de los seres humanos, y también en la producción animal.
Además, las especies de Salmonella tienen la capacidad de formar biopelícula, donde los microorganismos celulares están incrustados en una matriz extracelular, por lo que estos microorganismos pueden adherirse a superficies bióticas o abióticas (DAVEY y O’TOOLE, 2000; FLEMMING et al., 2016). Las matrices complejas de la biopelícula mantienen las células microbianas protegidas de la acción del proceso de saneamiento y los agentes antimicrobianos, por lo tanto, cuando los microorganismos patógenos están involucrados en la biopelícula, existe un gran riesgo de contaminar los alimentos, lo que conduce a un problema de salud pública. (KASNOWSKY et al., 2010).
Por lo tanto, el objetivo de este estudio fue caracterizar los determinantes de la resistencia a los antibióticos y evaluar la producción de biofilm en cepas de Salmonella spp. recuperadas de carne fresca de aves de corral vendida en una metrópoli brasileña.
MÉTODOS
Muestreo e identificación de las canales de aves de corral
Se compraron un total de 61 canales de carne de aves de corral en 07 mercados públicos diferentes en la ciudad de Recife, en el estado de Pernambuco, Brasil, entre 2018 y 2019 (figura 1 y tabla 1). Las muestras fueron enviadas al Laboratorio de Inspección de Carnes y Leche (LICAL), del Departamento de Medicina Veterinaria de la Universidade Federal Rural de Pernambuco (UFRPE).
En el laboratorio, la superficie de cada paquete de canales de aves de corral se limpió con un 70 por ciento de alcohol. Luego, se tomaron al azar 25 g de las canales y se colocaron en bolsas estériles (stomacher) individuales con 225 ml de agua de peptona tamponada (Kasvi, Brasil), y se homogeneizaron en stomacher durante 2 min (Kasvi, Brasil) y se incubaron a 37 ° C durante 24 horas para la etapa de preenriquecimiento. Para la etapa de enriquecimiento, 0,1 ml del caldo preenriquecido se transfirieron a 10 ml de caldo de tetrationato (Merck e pais) y luego se incubaron a 41,5 °C durante 24 horas, y 1,0 ml del caldo preenriquecido también se transfirieron a 10 ml de Rappaport y se incubaron a 37 °C durante 24 horas para el enriquecimiento selectivo. Después de la incubación, un bucle de caldo TT y cultivos de Rappaport se rayaron en placas de agar xilosa lisina (XLD) (Kasvi, Brasil) y agar entérico Hektoen (HE) (Kasvi, Brasil) e incubadas a 37 °C durante 24 h. Se seleccionaron tres presuntas colonias de Salmonella en las placas y se rayaron en agar nutriente (Merck, pais-) y se incubaron a 37,8 °C durante 24 horas. Luego, las colonias del agar nutriente fueron sometidas a pruebas bioquímicas y serológicas (ISO, 657912017).
Figura 1: Mercados públicos donde se adquirieron las muestras
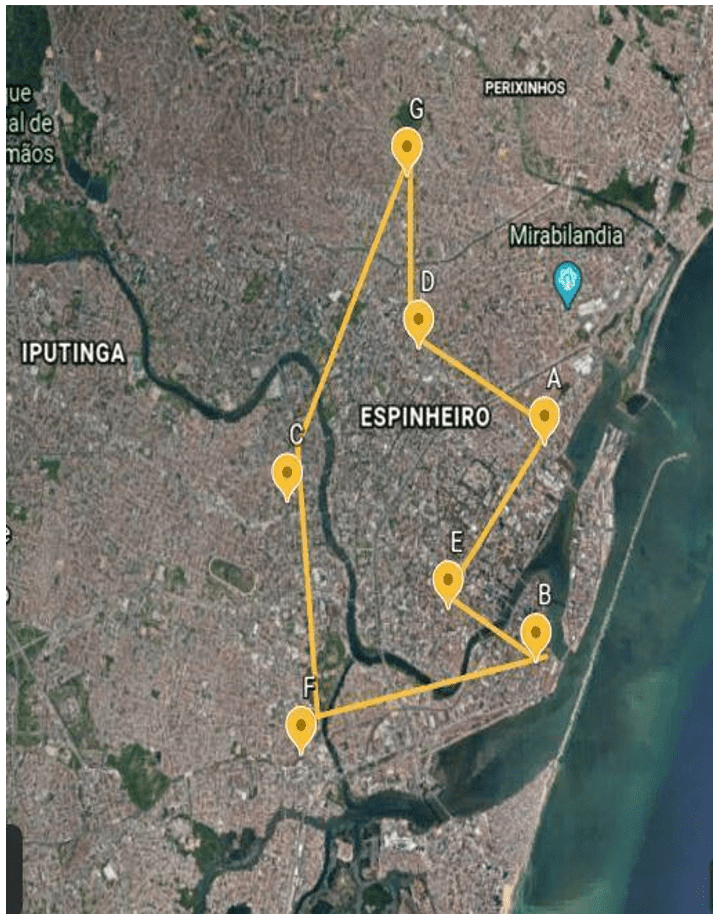
Tabla 1: Número de muestras adquiridas en diferentes mercados públicos de la ciudad de Recife – Pernambuco, Brasil
| Mercado público | Muestras adquiridas |
| Un | 1, 2, 3 22, 23, 24, 25, 26 |
| B | 4, 5, 6, 7, 8, 9, 27, 28, 29 |
| C | 30, 31, 32 |
| D | 10, 33, 34, 35, 36, |
| E | 11, 37, 38, 39, 40, 41 |
| F | 12, 13, 14, 15, 16, 17, 18, 45, 46,47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61 |
| G | 18, 20, 21, 42, 43, 44 |
Fuente: Elaboración propia.
Perfil de susceptibilidad a los antimicrobianos La prueba del perfil de susceptibilidad se realizó por el método de difusión del disco de agar con los siguientes agentes antimicrobianos: ampicilina, cloranfenicol, ciprofloxacina, ceftriaxona, sulfametoxazol-trimetoprima, imipenem, amoxicilina-ácido clavulánico, ceftazidima, cefotaxima y aztreonam, según el Instituto de Estándares Clínicos y de Laboratorio (CLSI) (CLSI, 2018).
Detección de genes de resistencia
El ADN utilizado en los análisis moleculares se obtuvo a partir de suspensiones bacterianas formadas por colonias frescas, que previamente se cultivaron durante 16-18 horas en agar Luria Bertani e inocularon en aproximadamente 300 μL de agua ultrapura libre de nucleasa homogeneizada con la ayuda de un agitador de tubo de vórtice (Vision Scientific).
La investigación de genes para la resistencia a β-lactámicos y quinolonas se realizó mediante PCR. Las condiciones de ciclo térmico fueron las descritas por los autores enumerados en la tabla de cebadores.
Tabla 2: Cartillas utilizadas para detectar los genes de las β-lactamasas
| Cebador | 5′ 3′ Secuencias | Gen diana | Producto | Referencias | |
| KPC | F | TGTCACTGTATCGCCGTC | blaKPC | 1011pb | (Bratu et al., 2005) |
| R | CTCAGTGCTCTACAGAAAACC | ||||
| CTX-M | F | SCSATGTGCAGYACCAGTAA | blaCTXM | 500pb | (Cao et al., 2002) |
| R | CCGCRATATGRTTGGTGGTG | ||||
| TEM | F | TCGGGGAAATGTGCGCG | blaTEM | 700pb | |
| R | TGCTTAATCAGTGAGGCACC | ||||
| SHV | F | TTATCTCCCTGTTAGCCACC | blaSHV | 900pb | |
| R | GATTTGCTGATTTCGCTCGG | ||||
| R | AAGCAGACTTGACCTGA | ||||
| qnrA | F | AGA GGA TTT CTC ACG CCA GG | qnrA | 580pb | (Cattoir et al., 2007). |
| R | TGC CAG GCA CAG ATC TTG AC | ||||
| qnrB | F | GGM ATH GAA ATT CGC CAC T | qnrB | 264pb | |
| R | TTT GCY GYY CGC CAG TCG AA | ||||
| qnrC | F | GGG TTG TAC ATT TAT TGA ATC | qnrC | 447PB | (Wang et al., 2009) |
| R | TCC ACT TTA CGA GGT TCT | ||||
| qnrD | F | CGA GAT CAA TTT ACG GGG AAT A | qnrD | 582pb | (Cavaco et al., 2009). |
Fuente: Elaboración propia.
Análisis del perfil genético de los aislados La comparación e identificación de variaciones genéticas en el contenido de cepas bacterianas se realizó mediante ERIC-PCR. Los cebadores utilizados para la reacción fueron ERIC-1R (5′ ATGTAAGCTCCTGGGGATTCAC-3′) y ERIC2 (5’AAGTAAGTGACTGGGGGGGGGCCG-3′) (Sigma Aldrich) según (VERSALOVIC et al., 1991), con la modificación descrita por (FENDRI et al., 2013).
Capacidad de producción de biofilm
La capacidad de formación de biofilm se evaluó de acuerdo con la metodología propuesta por (STEPANOVIĆ et al., 2000; STEPANOVIĆ et al., 2007). Cada cepa se diluyó a 108UFC/ml (0,5 en la escala de MacFarland) utilizando caldo de tripticasa de soja (TSB) (Merck), y se cultivaron 200 μL en tres pocillos de la microplaca de poliestireno de fondo plano de 96 pocillos (Nest®). Se utilizaron un total de 69 pozos para probar 21 cepas; los otros 3 pocillos recibieron el Salmonella Typhimurim ATCC 14028 como control positivo, y 3 pocillos recibieron el control negativo (medio de cultivo no inoculado). Las placas que contenían cepas de Salmonella spp. y controles se incubaron a 35ºC durante 96 horas. La placa se lavó tres veces con solución salina tamponada con fosfato (PBS pH 7.2) y se tiñó con violeta cristalina al 1% durante 15 minutos. Después de lavar tres veces con agua destilada y secar a temperatura ambiente, la absorbancia se leyó en un lector de microplacas Polaris (Celer®) a una longitud de onda de 492 nm (SERENO et al., 2017).
La densidad óptica (OD)[11] de cada cepa de Salmonella spp. se obtuvo por la media aritmética de la absorbancia de tres pocillos y este valor se comparó con la absorbancia media de los controles negativos (ODnc). Después de eso, las cepas se clasificaron en ningún productor de biofilm (OD≤ODnc); productor débil de biofilm (ODnc<ODs≤2.ODnc); productor moderado de biofilm (2.ODnc<ODs≤4.ODnc); y una fuerte clasificación de producción de biofilm (4.ODnc<ODs) dada de acuerdo con (STEPANOVIĆ et al., 200; STEPANOVIĆ et al., 2007).
RESULTADOS
Prevalencia de Salmonella spp. en muestras de canales de carne de aves de corral
De las 61 muestras de canales de carne de aves de corral recogidas en los mercados públicos, el 34% (21) estaban contaminadas por Salmonella spp. La prevalencia de Salmonella spp. en cada mercado público se expresa en la figura 3, y los datos se detallan en la tabla 3.
Figura 2: Prevalencia de aislados de Salmonella spp. de cada mercado público en la ciudad de Recife – Pernambuco, Brasil
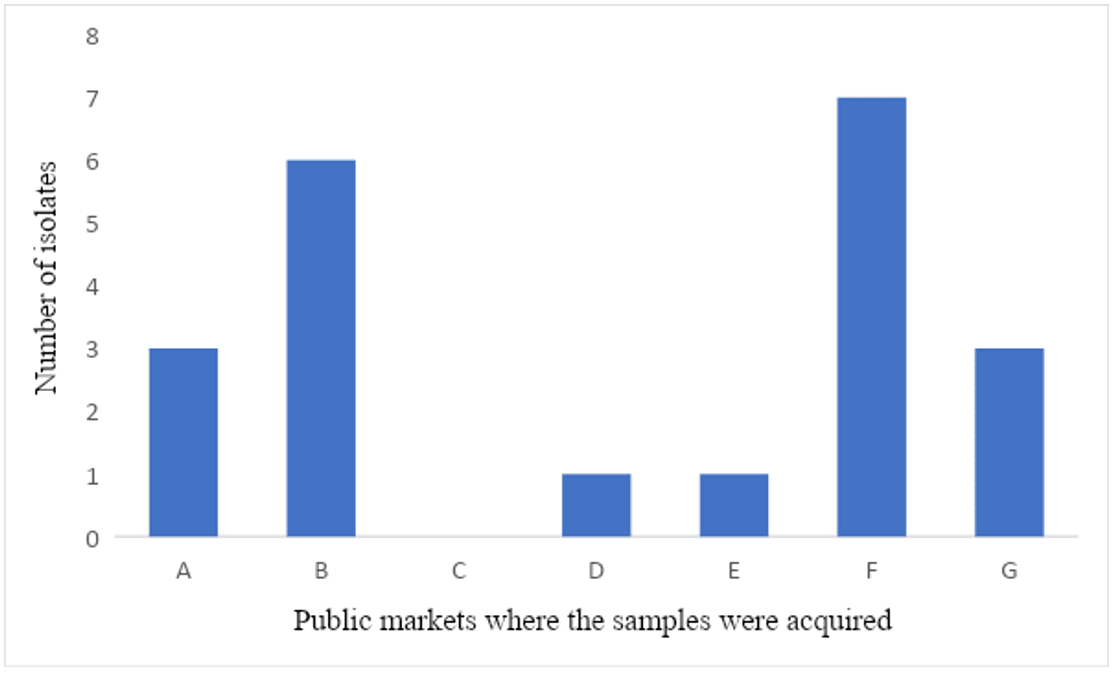
Tabla 3: Salmonella spp. aislada de cada mercado público en la ciudad de Recife – Pernambuco, Brasil
| Aislantes | Mercado público | Año |
| 1 | Un | 2018 |
| 2 | 2019 | |
| 3 | 2019 | |
| 4 | B | 2018 |
| 5 | 2018 | |
| 6 | 2018 | |
| 7 | 2018 | |
| 8 | 2018 | |
| 9 | 2018 | |
| 10 | D | 2019 |
| 11 | E | 2019 |
| 12 | F | 2019 |
| 13 | 2019 | |
| 14 | 2019 | |
| 15 | 2019 | |
| 16 | 2019 | |
| 17 | 2019 | |
| 18 | 2019 | |
| 19 | G | 2018 |
| 20 | 2019 | |
| 21 | 2019 |
Fuente: Elaboración propia.
Figura 3: Resistencia antimicrobiana a agentes antimicrobianos individuales entre aislados de Salmonella spp. de canales de aves de corral vendidas en mercados públicos en la ciudad de Recife – Brasil
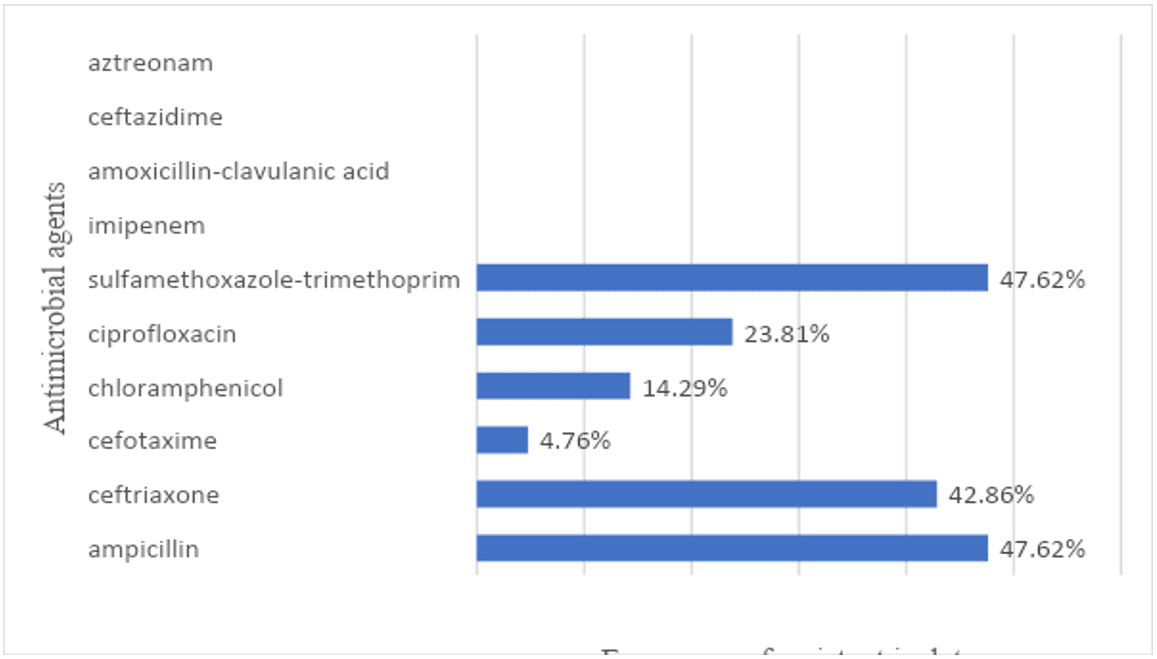
Perfil de susceptibilidad
De los 21 aislados, 13 (61,9%) fueron resistentes a al menos un agente antimicrobiano. La resistencia antimicrobiana a agentes antimicrobianos individuales entre los aislados de Salmonella se expresa en la figura.3. Los datos detallados de la prueba de sensibilidad antimicrobiana se expresan en la tabla 4.
Tabla 4. Perfil de susceptibilidad antimicrobiana de aislados de canales de pollo adquiridas en mercados públicos de la ciudad de Recife – PE, entre 2018 y 2019
| Aislar | AMPERIO | AMC | CRO | CTX | CAZ | IPM | CAJERO | CLO | CIP | SUT |
| 1 | R | S | R | S | S | S | S | S | R | R |
| 2 | S | S | S | S | S | S | S | S | Yo | S |
| 3 | S | S | S | S | S | S | S | S | R | R |
| 4 | R | S | Yo | S | S | S | S | S | S | S |
| 5 | S | S | S | S | S | S | S | S | Yo | S |
| 6 | S | S | Yo | S | S | S | S | S | R | S |
| 7 | S | S | Yo | S | S | S | S | S | Yo | S |
| 8 | S | S | S | S | S | S | S | S | R | S |
| 9 | S | S | Yo | S | S | S | S | S | Yo | S |
| 10 | S | S | S | S | S | S | S | S | Yo | S |
| 11 | S | S | S | S | S | S | S | S | Yo | S |
| 12 | R | S | R | S | S | S | S | S | Yo | R |
| 13 | R | S | R | S | S | S | S | S | Yo | R |
| 14 | S | S | S | S | S | S | S | S | Yo | S |
| 15 | R | S | R | S | S | S | S | R | S | R |
| 16 | R | S | R | S | S | S | S | R | S | R |
| 17 | R | S | R | S | S | S | S | R | S | R |
| 18 | R | S | R | S | S | S | S | S | S | R |
| 19 | S | S | S | S | S | S | S | S | S | S |
| 20 | R | S | R | S | S | S | S | S | S | R |
| 21 | R | S | R | R | S | S | S | S | R | R |
AMP – Ampicilina, CLO – cloranfenicol, CIP – ciprofloxacino, CRO- ceftriaxona, SUT – sulfametoxazol-trimetoprima, IPM – imipenem, AMC – amoxicilina-ácido clavulánico, CAZ – ceftazidima, CTX- cefotaxima y ATM – Aztreonam, S – sensible; R- Resistente; Yo-. Intermedio. Fuente: Elaboración propia.
Se identificaron al menos dos genes de resistencia en todos los aislamientos, entre los cuales se observó la presencia de genes de resistencia que codifican β-lactamasas (bla) y genes de resistencia que codifican quinolonas (qnr) en las pruebas moleculares realizadas en el presente estudio, y los resultados se detallan en la tabla 3.
Tabla 5: Genes de resistencia detectados en cada aislado de Salmonella spp, recuperados de canales de aves adquiridas en mercados públicos de la ciudad de Recife – Pernambuco entre los años 2018 y 2019
| Resistente a β-lactámicos | Resistentes a las quinolonas | |||||||
| Aislantes | BlaTEM-like | BlaSHV-like | BlaCTX-M-like | BlaKPC-2 | qnrA | qnrB | qnrC | qnrD |
| 1 | + | – | – | – | + | + | – | – |
| 2 | + | – | – | – | + | + | – | – |
| 3 | + | – | – | – | + | + | – | + |
| 4 | + | – | – | – | + | + | – | – |
| 5 | + | – | – | – | + | + | – | + |
| 6 | + | – | – | – | + | + | – | + |
| 7 | + | – | – | – | + | + | – | + |
| 8 | + | – | – | – | + | + | – | + |
| 9 | + | – | – | – | + | + | – | + |
| 10 | + | – | + | – | – | + | – | – |
| 11 | + | – | – | – | – | + | – | – |
| 12 | + | – | + | – | – | + | – | – |
| 13 | + | – | + | – | – | + | – | – |
| 14 | + | – | + | – | – | + | – | – |
| 15 | + | – | + | – | – | + | – | – |
| 16 | + | – | + | – | – | + | – | + |
| 17 | – | – | + | – | – | + | – | + |
| 18 | + | – | + | – | – | + | – | + |
| 19 | + | – | – | – | – | + | – | + |
| 20 | + | – | – | – | – | + | – | + |
| 21 | + | – | – | – | + | + | – | + |
Fuente: Elaboración propia.
Relación clonal Los aislados mostraron un patrón que varió de 3 a 9 tiras, lo que reveló la presencia de diferentes genotipos patrón indistinguibles entre sí y otros cuyo estándar es diferente para sólo una o dos bandas. Algunos aislados de Salmonella spp. mostraron patrones genéticos idénticos (2, 3, 4, 5, 6 y 7). Las muestras 9, 11, 19, 20 y 21 estaban estrechamente relacionadas genéticamente, ya que tienen un patrón similar de tiras, diferenciándose solo en una (9, 21) o dos tiras (11 y 20).
Formación de biofilm
Todos los 21 aislados de Salmonella spp. analizados eran productores de biofilms, y estos aislados se clasificaron como productores débiles de biofilm según (STEPANOVIĆ et al., 2000; STEPANOVIĆ et al., 2007).
DISCUSIÓN
Este trabajo identificó Salmonella spp. contaminando canales de pollo vendidas en mercados públicos en una metrópoli brasileña. Además, detectamos los mecanismos de resistencia presentes en estos aislados bacterianos y confirmamos que todos eran productores de biofilm. Los resultados que se presentan aquí mostraron varios aislados con una proximidad genética y revelaron que los otros son clones.
Los aislados 2, 3, 4 y 5 tienen un perfil genético idéntico, aunque se recolectaron en dos mercados diferentes, el Mercado A y el Mercado B, que están a unos 5 km de distancia entre sí. Este hallazgo indica que los vendedores pueden tener el mismo proveedor, donde comenzó la contaminación por Salmonella MDR. El mercado F presentó un mayor número de cepas caracterizadas como clones (12, 14, 15, 16 y 18). Además de la posibilidad del mismo proveedor, de acuerdo con un conjunto de fallas en las condiciones higiénicas, como la exposición de los productos al polvo y los insectos y el intercambio inadecuado de utensilios como cuchillos y contenedores entre los comerciantes, pueden contribuir a la contaminación de las canales, lo que resulta en la diseminación de aislados isogénicos (AZEVEDO, 2014).
La presencia de Salmonella spp. en la carne de aves de corral también se observó en los estudios realizados por (BAPTISTA et al., 2018; MELO et al., 2020; YIN et al., 2021). Sin embargo, nuestros hallazgos difieren de estos estudios debido a la mayor prevalencia de aislados de Salmonella spp.
La contaminación por Salmonella spp. detectada en las muestras puede deberse a una contaminación previa en el propio matadero, debido a fallos en el proceso de sacrificio (LOPES et al., 2007; VON RÜCKERT et al., 2009). También se observó que los comerciantes retiraron los cadáveres del embalaje original y los colocaron en recipientes de plástico y luego los vendieron a temperatura ambiente (88 ºF), lo que se considera un fraude. Esta temperatura promueve la multiplicación de Salmonella spp., ya que estos microorganismos pueden multiplicarse a temperaturas alrededor de 5 ° C a 45 ° C (MAHMOUD, 2012) Este problema relacionado con la temperatura también se notó en los estudios realizados por (KHAN et al., 2018; JARQUIN et al., 2015).
Por lo tanto, esta práctica aumenta el riesgo de contaminación del producto, principalmente debido a prácticas de manipulación inadecuadas y condiciones higiénicas sanitarias inadecuadas y a lo que se observa en estos mercados públicos, ya que se encontró que las mesas y bancos no se limpiaron en el 59% (36/61) de los lugares. En cuanto a los manipuladores, se observó que el 100% no tenía las uñas bien cortadas y limpias. Además, el mismo empleado que cuidaba la carne era el mismo en la caja. En consecuencia, estas fallas en los procesos de buenas prácticas de manejo observadas en estos mercados pueden contribuir a la contaminación y propagación de Salmonella spp. en este producto. Esta transferencia puede ocurrir entre varias interacciones entre el hombre, los alimentos y el medio ambiente. Por lo tanto, la contaminación cruzada es la base para la propagación de Salmonella spp.
A partir de los resultados de la prueba de susceptibilidad contra agentes antimicrobianos en el presente estudio, se observó que el 38% (08/21) de las cepas de Salmonella spp eran resistentes a múltiples fármacos (MDR)[12], es decir, bacterias que son resistentes a al menos tres clases de agentes antimicrobianos (MAGIORAKOS et al., 2011).
La aparición de cepas de Salmonella spp resistentes a múltiples antimicrobianos en carne de ave en Brasil es una realidad creciente, debido a los datos reportados en el presente estudio, y otras investigaciones realizadas por (ZIECH, 2015; BAPTISTA et al., 2018). Puede suceder debido al uso intensivo de agentes antimicrobianos en la producción animal para tratar a los animales, y también para mejorar la producción.
La mayor implicación en cuanto a la diseminación de cepas zoonóticas multirresistentes está relacionada con fallos terapéuticos frente a varias condiciones, tanto en casos humanos como animales. Además, esta realidad conlleva pérdidas en los rebaños de animales, ya que cuando están presentes, estas cepas resistentes son difíciles de eliminar. La permanencia de cepas patógenas y multirresistentes en la producción avícola es aún más preocupante, ya que la densidad animal es mayor y, en consecuencia, hay una mayor propagación de los microorganismos en un período de tiempo más corto.
Se observó que sólo el 4,28% (03/21) de los aislamientos eran resistentes al cloranfenicol. Este resultado puede reflejar la prohibición del uso de este agente como agente terapéutico y promotor del crecimiento en la producción animal en Brasil (PACHECO-SILVA et al., 2014).
Por otro lado, el 42,85% (09/21) de los aislados fueron resistentes a la ceftriaxona, cefalosporina de 3ª generación. El uso de este medicamento en la producción animal está prohibido en Brasil (MION et al., 2014). Por lo tanto, este resultado indica que este medicamento u otros de la misma clase de antimicrobianos todavía se utilizan en la producción animal en Brasil.
Además, 42,85 (9/21) de los aislados fueron resistentes a tres o más fármacos antimicrobianos. Luego, al comparar el perfil resistente con los mercados, se observó que los aislados número 13, 15, 16, 17, 18, que eran resistentes a hasta tres medicamentos eran del mismo mercado público (mercado público F). Sugiere que puede haber habido contaminación cruzada en el mercado, o que estas canales de pollo pueden haber sufrido contaminación previa en el matadero de aves de corral.
Por lo tanto, la presencia de cepas MDR de Salmonella en el origen animal de la alimentación está aumentando en Brasil (BAPTISTA et al., 2018; MELO et al., 2020; VOSS-RECH et al., 2015). Las infecciones relacionadas con cepas multirresistentes se asocian con una alta morbilidad y mortalidad, en comparación con las sensibles, ya que estos microorganismos representan una barrera en el tratamiento de enfermedades humanas y animales.
La presencia de tales genes de resistencia detectados en aislados de Salmonella spp. de alimentos de origen animal representa una seria amenaza para la salud pública, ya que la transmisión horizontal de genes de resistencia ocurre principalmente por plásmidos que codifican β-lactamasas (GYLES, 2008). Además, estos genes se consideran de origen comunitario y son comunes en entornos hospitalarios (SUN et al., 2013). Así, la diseminación de cepas resistentes señala la importancia del control de Salmonella spp en la carne de ave (BORGES et al., 2019; SIVASANKAR et al., 2020).
La presencia de cepas formadoras de biofilm de Salmonella spp. observada en el presente estudio señala el riesgo que este hallazgo puede traer a la industria alimentaria y la salud pública. La capacidad de formar biopelícula protegió a los microorganismos dentro de la biopelícula y, por lo tanto, menos susceptibles a factores externos, como la acción de agentes antimicrobianos (ZIECH, 2015; SERENO et al., 2017; BORGES et al., 2018).
Además, las biopelículas promueven la permanencia de microorganismos patógenos y de deterioro, que causan enfermedades a los consumidores, y la depreciación del producto final debido a cambios físico-químicos y sensoriales (KASNOWSKY et al., 2010; SINGH et al., 2017).
Finalmente, esta investigación mostró que la identificación de Salmonella spp. productora de biofilm y portadora de diferentes genes de β-lactamasa y determinantes de resistencia a quinolonas demuestra la capacidad de estas bacterias para acumular diversos mecanismos de virulencia y resistencia a antimicrobianos. La presión selectiva que ejerce el uso indiscriminado de antibióticos en la agricultura es un factor importante para la adquisición de diferentes mecanismos de resistencia y diseminación de estas cepas. Además, la propagación de diferentes grupos clonales de Salmonella spp. MDR, en canales de pollo, mostrada en este estudio atestigua la necesidad de controles efectivos para contener este microorganismo, que además de ser un riesgo para la salud pública, también es responsable de considerables pérdidas económicas.
APOYO FINANCIERO
Los autores agradecen a la Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) por la beca otorgada.
REFERENCIAS
ALEKSHUN, M. N.; LEVY, S. B. Molecular Mechanisms of Antibacterial Multidrug Resistance. Cell, v. 128, n. 6, p. 1037–1050, 23 mar. 2007. Available from: https://doi.org/10.1016/j.cell.2007.03.004. Acessed in: 02 mar. 2021.
AZEVEDO, S. M. M. Farmacologia dos antibióticos beta-lactâmicos. Porto, Portugal: Universidade Fernando Pessoa, 2014. Available from: https://bdigital.ufp.pt/handle/10284/4412. Acessed in: 02 mar. 2021.
BAPTISTA, D. Q. et al. Prevalência e susceptibilidade antimicrobiana de sorotipos de Salmonella spp. isolados de frangos vivos e carcaças no estado do Rio de Janeiro. Pesquisa Veterinária Brasileira, v. 38, n. 7, p. 1278–1285, jul. 2018. Available from: https://doi.org/10.1590/1678-5150-PVB-5289. Acessed in: 02 mar. 2021.
BORGES, K. A. et al. Biofilm formation capacity of Salmonella serotypes at different temperature conditions. Pesquisa Veterinária Brasileira, v. 38, n. 1, p. 71–76, jan. 2018. Available from: https://doi.org/10.1590/1678-5150-pvb-4928. Acessed in: 02 mar. 2021.
BORGES, K. A.; MARTELO, E. B.; DOS SANTOS, L. A.; FURIAN, T. Q.; CISCO, I. C.; MANTO, L.; DOS SANTOS, L. R. Detection and quantification of Salmonella spp. in poultry slaughterhouses of southern Brazil. The Journal of Infection in Developing Countries, [S. l.], v. 13, n. 05, p. 455–460, 2019. DOI: 10.3855/jidc.11107. Available from: https://jidc.org/index.php/journal/article/view/32053516. Acessed in: 02 mar. 2021.
BRATU, S.; LANDMAN, D.; ALAM, M.; TOLENTINO, E.; QUALE, J. Detection of KPC Carbapenem-Hydrolyzing Enzymes in Enterobacter spp. from Brooklyn, New York. Antimicrobial Agents and Chemotherapy, v. 49, n. 2, p. 776–778, fev. 2005. Available from: https://doi.org/10.1128/AAC.49.2.776-778.2005. Acessed in: 02 mar. 2021.
BRASIL. Manual Integrado de Vigilância, Prevenção e Controle de Doenças Transmitidas por Alimentos. Ministério da Saúde, Secretária de Vigilância em Saúde, 2019. Available from: http://portalarquivos2.saude.gov.br/images/pdf/2019/fevereiro/15/Apresenta oSurtos-DTA—Fevereiro-2019.pdf. Acessed in: 13 Aug. 2019.
BONI, H. F. K.; CARRIJO, A. S.; FASCINA, V. B. Detection of Salmonella spp. in broiler buildings and stuff of slaughterhouse in the central region of Mato Grosso do Sul. R. bras. Saúde Prod. Anim., 2011. Available from: http://revistas.ufba.br/index.php/rbspa/article/view/1949. Acessed in: 02 mar. 2021.
CAO, V.; LAMBERT, T.; NHU, D. Q.; LOAN, H. K.; HOANG, N. K.; ARLET, G.; COURVALIN, P. Distribution of Extended-Spectrum β-Lactamases in Clinical Isolates of Enterobacteriaceae in Vietnam. Antimicrobial Agents and Chemotherapy, v. 46, n. 12, p. 3739–3743, dez. 2002. Available from: https://doi.org/10.1128/AAC.46.12.3739-3743.2002. Acessed in: 02 mar. 2021.
CATTOIR, V.; POIREL, L.; ROTIMI, V.; SOUSSY, C. J.; NORDMANN, P. Multiplex PCR for detection of plasmid-mediated quinolone resistance qnr genes in ESBL-producing enterobacterial isolates. Journal of Antimicrobial Chemotherapy, v. 60, n. 2, p. 394–397, 1 ago. 2007. Available from: https://doi.org/10.1093/jac/dkm204. Acessed in: 02 mar. 2021.
CAVACO, L. M.; HASMAN, H.; XIA, S.; AARESTRUP, F. M. qnrD, a novel gene conferring transferable quinolone resistance in Salmonella enterica serovar Kentucky and Bovismorbificans strains of human origin. Antimicrobial Agents and Chemotherapy, v. 53, n. 2, p. 603–608, fev. 2009. Available from: https://doi.org/10.1128/AAC.00997-08. Acessed in: 02 mar. 2021.
CENTRES FOR DISEASES, CONTROL AND PREVENTION – CDC. Salmonella. Centres For Diseases, Control And Prevention, 2020. Available from: https://www.cdc.gov/salmonella/index.html. Acessed in: 02 mar. 2021.
DAVEY, M. E.; O’TOOLE, G. A. Microbial Biofilms: from Ecology to Molecular Genetics. Microbiology and Molecular Biology Reviews, v. 64, n. 4, p. 847–867, dez. 2000. Available from: https://doi.org/10.1128/mmbr.64.4.847-867.2000. Acessed in: 02 mar. 2021.
DUARTE, D. A. M. et al. Occurrence of Salmonella spp. in broiler chicken carcasses and their susceptibility to antimicrobial agents. Brazilian Journal of Microbiology, v. 40, n. 3, p. 569–573, set. 2009. Available from: https://doi.org/10.1590/S1517-83822009000300020. Acessed in: 02 mar. 2021.
FENDRI, I.; BEN HASSENA, A.; GROSSET, N.; BARKALLAH, M.; KHANNOUS, L.; CHUAT, V.; GAUTIER, M.; GDOURA, R. Genetic Diversity of Food-Isolated Salmonella Strains through Pulsed Field Gel Electrophoresis (PFGE) and Enterobacterial Repetitive Intergenic Consensus (ERIC-PCR). PLOS ONE, v. 8, n. 12, p. e81315, 3 dez. 2013. Available from: https://doi.org/10.1371/journal.pone.0081315. Acessed in: 02 mar. 2021.
FITCH, F. M.; CARMO-RODRIGUES, M. S.; OLIVEIRA, V. G. S.; GASPARI, M. V.; DOS SANTOS, A.; DE FREITAS, J. B.; PIGNATARI, A. C. C. β-Lactam Resistance Genes: Characterization, Epidemiology, and First Detection of blaCTX-M-1 and blaCTX-M-14 in Salmonella spp. Isolated from Poultry in Brazil—Brazil Ministry of Agriculture’s Pathogen Reduction Program. Microbial Drug Resistance, v. 22, n. 2, p. 164–171, 8 mar. 2016. Available from: https://doi.org/10.1089/mdr.2015.0143. Acessed in: 02 mar. 2021.
FLEMMING, H. C.; WINGENDER, J.; SZEWZYK, U.; STEINBERG, P.; RICE, S. A.; KJELLEBERG, S. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology, v. 14, n. 9, p. 563–575, 11 ago. 2016. Available from: https://doi.org/10.1038/nrmicro.2016.94. Acessed in: 02 mar. 2021.
GRIMONT, P.; WEILL, F. X. Antigenic formulae of the Salmonella servovars: WHO Collaborating Centre for Reference and Research on Salmonella. 9th Ed. Inst. Pasteur, 2007. 1–166 p.
GYLES, C. L. Antimicrobial resistance in selected bacteria from poultry. Animal Health Research Reviews, v. 9, n. 2, p. 149–158, 2008. Available from: https://doi.org/10.1017/S14662523080015522. Acessed in: 02 mar. 2021.
JARQUIN, C.; ALVAREZ, D.; MORALES, O.; MORALES, A. J.; LÓPEZ, B.; DONADO, P.; VALENCIA, M. F.; ARÉVALO, A.; MUÑOZ, F.; WALLS, I.; DOYLE, M. P.; ALALI, W. Q. Salmonella on Raw Poultry in Retail Markets in Guatemala: Levels, Antibiotic Susceptibility, and Serovar Distribution. Journal of Food Protection, v. 78, n. 9, p. 1642–1650, set. 2015. Available from: https://doi.org/10.4315/0362-028X.JFP-15-117. Acessed in: 02 mar. 2021.
KASNOWSKY, M. C. et al. Formação de biofilme na indústria de alimentos e métodos de validação de superfícies. Revista Científica Eletrônica de Medicina Veterinária, v. 15, p. 1679–7353, jul. 2010. Available from: http://faef.revista.inf.br/imagens_arquivos/arquivos_destaque/fxPTiYWerLkT9Si_2013-6-25-16-32-0.pdf. Acessed in: 02 mar. 2021.
KHAN, A. S.; GEORGES, K.; RAHAMAN, S.; ABDELA, W.; ADESIYUN, A. A. Prevalence and serotypes of Salmonella spp. on chickens sold at retail outlets in Trinidad. PLOS ONE, v. 13, n. 8, p. e0202108, ago. 2018. Available from: https://doi.org/10.1371/journal.pone.0202108. Acessed in: 02 mar. 2021.
LOPES, M.; GALHARDO, J. A.; DE OLIVEIRA, T.; TAMANINI, R.; SANCHES, F.; MULLER, E. Pesquisa de Salmonella spp. e microrganismos indicadores em carcaças de frango e água de tanques de pré-resfriamento em abatedouro de aves. Semina: Ciências Agrárias, v. 28, n. 3, p. 465–476, 30 ago. 2007. Available from: https://doi.org/10.5433/1679-0359.2007v28n3p465. Acessed in: 02 mar. 2021.
MAGIORAKOS, A. P.; SRINIVASAN, A.; CAREY, R. B.; CARMELI, Y.; FALAGAS, M. E.; GISKE, C. G.; HARBARTH, S.; HINDLER, J. F.; KAHLMETER, G.; OLSSON-LILJEQUIST, B.; PATERSON, D. L.; RICE, L. B.; STELLING, J.; STRUELENS, M. J.; VATOPOULOS, A.; WEBER, J. T.; MONNET, D. L. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clinical microbiology and infection: the official publication of the European Society of Clinical Microbiology and Infectious Diseases, v. 18, n. 3, p. 268–281, 2012. Available from: https://pubmed.ncbi.nlm.nih.gov/21793988/. Acessed in: 02 mar. 2021.
MAHMOUD, B. S. M. Salmonella – A Dangerous Foodborne Pathogen, Nursing times, 2012. Available from: https://doi.org/10.1049/cm.1989.0039. Acessed in: 02 mar. 2021.
MELO, R. T. et al. Salmonella Minnesota de origem avícola apresenta fatores de virulência e risco potencial aos humanos. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v. 72, n. 4, p. 1353–1362, jul. 2020. Available from: https://doi.org/10.1590/1678-4162-10884. Acessed in: 02 mar. 2021.
MION, L.; COLLA, F. L.; CISCO, I. C.; WEBBER, B.; DIEDRICH, L. N.; PILOTTO, F.; RODRIGUES, L. B.; NASCIMENTO, V. P. do; SANTOS, L. R. dos. Perfil de resistência a antimicrobianos por Salmonella Heidelberg isoladas de abatedouro avícola em 2005 e 2009. Acta Scientiae Veterinariae, v. 42, n. 1, p. 1–5, 2014. Available from: https://www.redalyc.org/articulo.oa?id=289029240017. Acessed in: 02 mar. 2021.
PACHECO-SILVA, É.; SOUZA, J. R. de; CALDAS, E. D. Resíduos de medicamentos veterinários em leite e ovos. Química Nova, v. 37, n. 1, p. 111–122, 2014. Available from: https://doi.org/10.1590/S0100-40422014000100020. Acessed in: 02 mar. 2021.
PENG, M.; SALAHEEN, S.; BISWAS, D. Animal Health: Global Antibiotic Issues. Encyclopedia of Agriculture and Food Systems, p. 346–357, jan. 2014. Available from: https://doi.org/10.1016/B978-0-444-52512-3.00187-X. Acessed in: 02 mar. 2021.
REZENDE, C. S. M. e; MESQUITA, A. J. de; ANDRADE, M. A.; LINHARES, G. F. C.; MESQUITA, A. Q. de; MINAFRA, C. S. Sorovares de Salmonella isolados de carcaças de frangos de corte abatidos no Estado de Goiás, Brasil, e perfil de resistência a antimicrobianos. Revista Portuguesa de Ciências Veterinárias, Lisboa, v. 100, n. 555-556, p. 199-203, jul./dez. 2005. Available from: http://repositorio.bc.ufg.br/handle/ri/12309. Acessed in: 02 mar. 2021.
RIBEIRO, A. R. et al. Salmonella spp. in raw br
oiler parts: occurrence, antimicrobial resistance profile and phage typing of the Salmonella Enteritidis isolates. Brazilian Journal of Microbiology, v. 38, n. 2, p. 296–299, abr. 2007. Available from: https://doi.org/10.1590/S1517-83822007000200021. Acessed in: 02 mar. 2021.
SERENO, M. et al. Antimicrobial Susceptibility and Biofilm Production by Salmonella sp. Strains Isolated from Frozen Poultry Carcasses. Brazilian Journal of Poultry Science, v. 19, n. 1, p. 103–108, jan. 2017. Available from: https://doi.org/10.1590/1806-9061-2016-0268. Acessed in: 02 mar. 2021.
SINGH, S.; SINGH, S. K.; CHOWDHURY, I.; SINGH, R. Understanding the Mechanism of Bacterial Biofilms Resistance to Antimicrobial Agents. The Open Microbiology Journal, v. 11, n. 1, p. 53–62, mai. 2017. Available from: https://doi.org/10.2174/1874285801711010053. Acessed in: 02 mar. 2021.
SIVASANKAR, C.; JHA, N. K.; GHOSH, R.; SHETTY, P. H. Anti quorum sensing and anti virulence activity of tannic acid and it’s potential to breach resistance in Salmonella enterica Typhi / Paratyphi A clinical isolates. Microbial Pathogenesis, v. 138, p. 103813, 1 jan. 2020. Available from: https://doi.org/10.1016/j.micpath.2019.103813. Acessed in: 02 mar. 2021.
STEPANOVIĆ, S.; VUKOVIĆ, D.; DAKIĆ, I.; SAVIĆ, B.; ŠVABIĆ-VLAHOVIĆ, M. A modified microtiter-plate test for quantification of staphylococcal biofilm formation. Journal of Microbiological Methods, v. 40, n. 2, p. 175–179, 1 abr. 2000. Available from: https://doi.org/10.1016/S0167-7012(00)00122-6. Acessed in: 02 mar. 2021.
STEPANOVIĆ, S.; VUKOVIĆ, D.; HOLA, V.; DI BONAVENTURA, G.; DJUKIĆ, S.; ĆIRKOVIĆ, I.; RUZICKA, F. Quantification of biofilm in microtiter plates: overview of testing conditions and practical recommendations for assessment of biofilm production by staphylococci. APMIS: acta pathologica, microbiologica, et immunologica Scandinavica, v. 115, n. 8, p. 891–899, 2007. Available from: https://pubmed.ncbi.nlm.nih.gov/17696944/. Acessed in: 02 mar. 2021.
SUN, D. D.; MA, X. X.; HU, J.; TIAN, Y.; PANG, L.; SHANG, H.; CUI, L. Z. Epidemiological and molecular characterization of community and hospital acquired Staphylococcus aureus strains prevailing in Shenyang, Northeastern China. The Brazilian Journal of Infectious Diseases, v. 17, n. 6, p. 682–690, 1 nov. 2013. Available from: https://doi.org/10.1016/j.bjid.2013.02.007. Acessed in: 02 mar. 2021.
THRELFALL, E. J. Antimicrobial drug resistance in Salmonella: problems and perspectives in food- and water-borne infections. FEMS Microbiology Reviews, v. 26, n. 2, p. 141–148, 1 jun. 2002. Available from: https://academic.oup.com/femsre/article/26/2/141/653178. Acessed in: 02 mar. 2021.
VERSALOVIC, J.; KOEUTH, T.; LUPSKI, R. Distribution of repetitive DNA sequences in eubacteria and application to finerpriting of bacterial enomes. Nucleic Acids Research, v. 19, n. 24, p. 6823–6831, dez. 1991. Available from: https://doi.org/10.1093/nar/19.24.6823. Acessed in: 02 mar. 2021.
VOSS-RECH, D.; VAZ, C. S. L.; ALVES, L.; COLDEBELLA, A.; LEAO, J. A.; RODRIGUES, D. P.; BACK, A. A temporal study of Salmonella enterica serotypes from broiler farms in Brazil. Poultry Science, v. 94, n. 3, p. 433–441, 1 mar. 2015. Available from: https://doi.org/10.3382/ps/peu081. Acessed in: 02 mar. 2021.
VON RÜCKERT, D. A. S. et al. Pontos críticos de controle de Salmonella spp. no abate de frangos. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v. 61, n. Arq. Bras. Med. Vet. Zootec., vol. 61, n. 2, p. 326–330, abr. 2009. Available from: https://doi.org/10.1590/S0102-09352009000200007. Acessed in: 02 mar. 2021.
WANG, M.; GUO, Q.; XU, X.; WANG, X.; YE, X.; WU, S.; HOOPER, D. C.; WANG, M. New plasmid-mediated quinolone resistance gene, qnrC, found in a clinical isolate of Proteus mirabilis. Antimicrobial Agents and Chemotherapy, v. 53, n. 5, p. 1892–1897, maio 2009. Available from: https://doi.org/10.1128/AAC.01400-08. Acessed in: 02 mar. 2021.
YIN, X.; M’IKANATHA, N. M.; NYIRABAHIZI, E.; MCDERMOTT, P. F.; TATE, H. Antimicrobial resistance in non-Typhoidal Salmonella from retail poultry meat by antibiotic usage-related production claims – United States, 2008–2017. International Journal of Food Microbiology, v. 342, p. 109044, 16 mar. 2021. Available from: https://doi.org/10.1016/j.ijfoodmicro.2021.109044. Acessed in: 02 mar. 2021.
ZIECH, R. E. Caracterização de Salmonella sp. isolada de indústrias de aves baseada na formação de biofilmes, tolerância a sanitizantes e resistência a antimicrobianos. Dissertação (Mestrado em Ciência Animal) – Programa de Pós-Graduação em Ciência Animal, Universidade Federal do Paraná. Palotina, 2015.
APÉNDICE – NOTA AL PIE
11. Optical Density (OD).
12. Microbial Drug Resistance (MDR).
[1] Estudiante de doctorado del Programa de Posgrado en Biociencia Animal. ORCID: 0000-0003-0698-6125. CURRÍCULO LATTES: http://lattes.cnpq.br/5170743479462841.
[2] Estudiante de doctorado del Programa de Posgrado en Biociencia Animal.ORCID: 0000-0001-9410-7631. CURRÍCULO LATTES: http://lattes.cnpq.br/4532231119888940.
[3] Doctor en Genética. ORCID: 0000-0003-0698-6125. CURRÍCULO LATTES: http://lattes.cnpq.br/4891800920829895.
[4] Licenciado en Veterinaria. ORCID: 0000-0001-9273-5204. CURRÍCULO LATTES: http://lattes.cnpq.br/8329028352662293.
[5] M.Sc. en Biociencia Animal. ORCID: 0000-0002-4913-8313. CURRÍCULO LATTES: http://lattes.cnpq.br/4967459162060058.
[6] Máster en Biología Celular y Molecular Aplicada. ORCID: 0000-0001-7764-0573. CURRÍCULO LATTES: http://lattes.cnpq.br/6281410502740086.
[7] Licenciado en Ciencias Biológicas.ORCID: 0000-0001-7928-7435. CURRÍCULO LATTES: http://lattes.cnpq.br/9549169192404311.
[8] Doctor en Bioquímica y Fisiología, Máster en Fisiología, Biólogo. ORCID: 0000-0003-1493-7964. CURRÍCULO LATTES: http://lattes.cnpq.br/9044747136928972.
[9] Doctor. ORCID: 0000-0002-1993-0350.
[10] Consejero. Doctor del Programa de Posgrado en Biociencia Animal. ORCID: 0000-0002-1289-2902. CURRÍCULO LATTES: http://lattes.cnpq.br/5998863169551704.
Enviado: 27 de marzo de 2023.
Aprobado: 08 de abril de 2023.