ARTIGO ORIGINAL
MELO, Nataly Sayonara da Silva [1], SILVA, Maria Goretti Varejão da [2], ALMEIDA, Anna Carolina Soares [3], MEDEIROS, Anna Karolyne de Araujo [4], SILVA, Daniel Dias da [5], SOUZA, Paula Mariana Salgueiro de [6], SILVA, Marcela Oliveira da [7], SOARES, Anísio Francisco [8], MENDONÇA, Marcelo [9], MEDEIROS, Elizabeth Sampaio de [10]
MELO, Nataly Sayonara da Silva, et al. Salmonela spp. multidroga virulenta e resistente recuperada de carcaças de frango no Brasil. Revista Científica Multidisciplinar Núcleo do Conhecimento. Ano. 08, Ed. 04, Vol. 01, pp. 92-114. Abril de 2023. ISSN: 2448-0959, Link de acesso: https://www.nucleodoconhecimento.com.br/biologia/salmonela-spp, DOI: 10.32749/nucleodoconhecimento.com.br/biologia/salmonela-spp
RESUMO
O objetivo deste trabalho foi avaliar a produção de biofilme, o perfil de suscetibilidade e a detecção de genes de resistência presentes em isolados de Salmonella spp de carcaças frescas de frango comercializadas em uma metrópole brasileira. De um total de 61 amostras de carcaças frescas de aves, 21 foram positivas para a presença de Salmonella spp. Em relação ao teste de suscetibilidade aos antimicrobianos, (13/21) os isolados testados foram resistentes a pelo menos um antibiótico, correspondendo a 61,9%, e 38% (21/08) foram resistentes a múltiplas drogas. Pelo menos dois genes de resistência foram identificados em todos os isolados, especialmente os genes relacionados às β-lactamases e à resistência às quinolonas. Observou-se também que alguns isolados de Salmonella spp apresentaram padrões genéticos idênticos. E todos os 21 isolados foram capazes de formar biofilme. A identificação do biofilme de Salmonella spp. formando e transportando diferentes genes β-lactamase e determinantes da resistência às quinolonas demonstra a capacidade dessas bactérias de acumular vários mecanismos de virulência e resistência aos antimicrobianos. Portanto, a disseminação de diferentes grupos clonais de Salmonella spp. MDR em carcaças de carne de aves de capoeira expressas neste atestam a necessidade de controles efetivos para conter esse microrganismo, que além de ser um risco para a saúde pública, também é responsável por perdas econômicas consideráveis.
Palavra-chave: Beta-lactâmicos, Intoxicação alimentar, PCR, Mercado público.
INTRODUÇÃO
A salmonelose é uma doença de origem alimentar causada por cepas patogênicas de Salmonella spp. Casos de salmonelose em humanos ocorrem principalmente através do consumo de alimentos ou água contaminados. Na maioria dos casos, os alimentos de origem animal, especialmente os de aves de capoeira, são as principais fontes de infecções por Salmonella spp. Os sintomas da infecção entérica são náuseas, vômitos, diarreia não sanguinolenta, febre, resfriado, dor abdominal, mialgia e dores de cabeça, e em pacientes imunodeprimidos pode levar a bacteremia, endocardite e morte (GRIMONT e WEILL, 2007).
De acordo com os Centros de Doenças, Controle e Prevenção (CDC), 1,2 milhão de casos de infecções, 23.000 hospitalizações e 450 mortes por ano nos Estados Unidos da América são causadas por Salmonella spp, aproximadamente. E as infecções relacionadas à ingestão de alimentos contaminados representam 1,0 milhão dos casos, aproximadamente (CDC, 2020). No Brasil, segundo o Ministério da Saúde, um total de 12.660 casos de doenças transmitidas por alimentos foi notificado entre 2000 e 2017, sendo a Salmonella spp. um dos principais agentes notificados nesses casos, o que representa um total de 35% dos casos notificados (BRASIL, 2019).
Na maioria dos casos de salmonelose, indivíduos saudáveis não precisam de tratamento. No entanto, pacientes imunodeprimidos precisam passar por tratamento (SERENO et al., 2017). No entanto, o uso indiscriminado de antimicrobianos, especialmente na produção animal, tem levado a um aumento do número de microrganismos resistentes a agentes terapêuticos, levando a um número limitado de opções antimicrobianas, o que pode resultar em falhas no tratamento (ALEKSHUN e LEVY, 2007; PENG et al., 2014). Ao longo dos anos, houve um aumento na prevalência de cepas de Salmonella resistentes a múltiplas drogas, principalmente em produtos avícolas, como a carne de frango (AZEVEDO, 2014; BAPTISTA et al., 2018; DUARTE et al., 2009; FITCH et al., 2016; BONI et al., 2011; REZENDE et al., 2005; RIBEIRO et al., 2007; THRELFALL, 2002).
Portanto, o surgimento da Salmonella multirresistente (MDR) é uma preocupação mundial, devido ao aumento das taxas de hospitalização e óbito. Este fenômeno é uma consequência do uso extensivo de antibióticos por seres humanos, e também na produção animal.
Além disso, as espécies de Salmonella têm a capacidade de formar biofilme, onde os microrganismos celulares são incorporados em uma matriz extracelular, de modo que esses microrganismos podem aderir a superfícies bióticas ou abióticas (DAVEY e O’TOOLE, 2000; FLEMMING et al., 2016). As matrizes complexas do biofilme mantêm as células microbianas protegidas da ação do processo de saneamento e agentes antimicrobianos, portanto, quando microrganismos patogênicos estão envolvidos no biofilme, há um enorme risco de contaminação do alimento, o que leva a um problema de saúde pública. (KASNOWSKY et al., 2010).
Assim, o objetivo deste estudo foi caracterizar os determinantes da resistência aos antibióticos e avaliar a produção de biofilme em cepas de Salmonella spp. recuperadas de carne fresca de aves de capoeira comercializada em uma metrópole brasileira.
MÉTODOS
Amostragem e identificação das carcaças de aves
Um total de 61 carcaças de carne de aves foi adquirido em 07 diferentes mercados públicos da cidade de Recife, no estado de Pernambuco, Brasil, entre 2018 e 2019 (figura 1 e tabela 1). As amostras foram encaminhadas ao Laboratório de Inspeção de Carnes e Leite (LICAL), do Departamento de Medicina Veterinária da Universidade Federal Rural de Pernambuco (UFRPE).
No laboratório, a superfície de cada embalagem de carcaças de aves foi limpa com 70% de álcool. Em seguida, 25g das carcaças foram colhidas aleatoriamente e colocadas em bolsas estéreis (stomacher) individuais com 225 ml de Água de Peptona Tamponada (Kasvi, Brasil), homogeneizadas em stomacher por 2 min (Kasvi, Brasil) e incubadas a 37 °C por 24 horas para a fase de pré-enriquecimento. Para a fase de enriquecimento, 0,1 ml do caldo pré-enriquecido foi transferido para 10 ml de caldo de tetrationato (Merck e pais) e depois incubado a 41,5 °C por 24 horas, e 1,0 ml do caldo pré-enriquecido também foi transferido para 10 ml de Rappaport e incubado a 37 °C por 24 horas para enriquecimento seletivo. Após a incubação, uma alça das culturas de caldo TT e Rappaport foi riscada nas placas de Ágar Lisina Xilose (XLD) (Kasvi, Brasil) e Ágar Entérico Hektoen (HE) (Kasvi, Brasil) e incubada a 37 °C por 24h. Três colônias presuntivas de Salmonella nas placas foram selecionadas e amarradas em ágar Nutrient (Merck, pais-) e incubadas a 37,8 °C por 24 horas. Em seguida, as colônias do ágar Nutrient foram submetidas a testes bioquímicos e sorológicos (ISO, 657912017).
Figura 1: Mercados públicos onde as amostras foram adquiridas
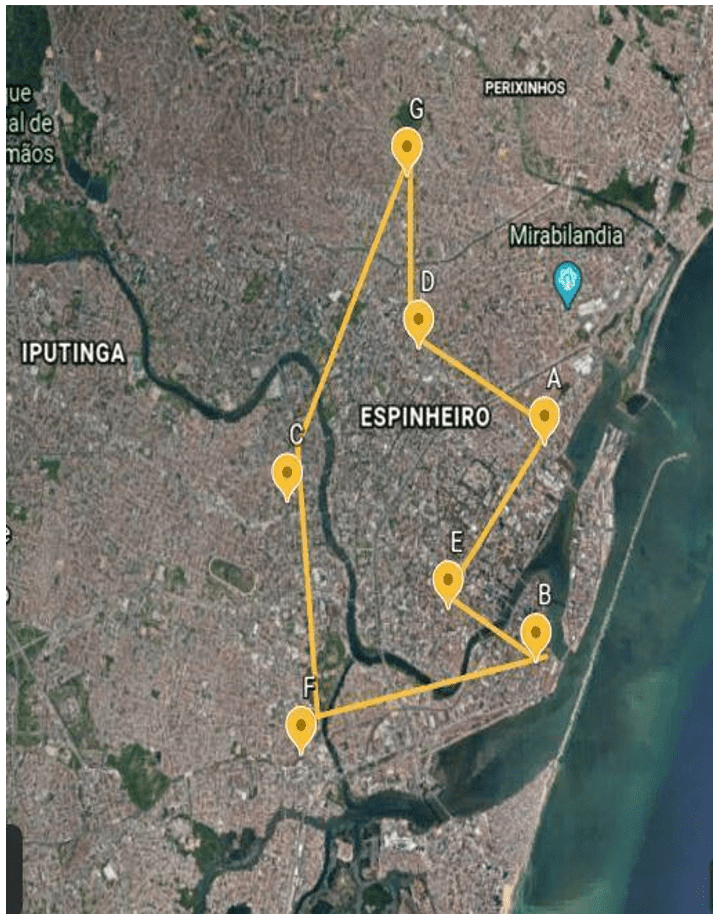
Tabela 1: Número de amostras adquiridas em diferentes mercados públicos da cidade do Recife – Pernambuco, Brasil
| Mercado Público | Amostras adquiridas |
| Um | 1, 2, 3 22, 23, 24, 25, 26 |
| B | 4, 5, 6, 7, 8, 9, 27, 28, 29 |
| C | 30, 31, 32 |
| D | 10, 33, 34, 35, 36, |
| E | 11, 37, 38, 39, 40, 41 |
| F | 12, 13, 14, 15, 16, 17, 18, 45, 46,47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61 |
| G | 18, 20, 21, 42, 43, 44 |
Fonte: Autores.
Perfil de suscetibilidade aos antimicrobianos
O teste do perfil de suscetibilidade foi feito pelo método de difusão em disco de ágar com os seguintes agentes antimicrobianos: ampicilina, cloranfenicol, ciprofloxacina, ceftriaxona, sulfametoxazol-trimetoprim, imipenem, amoxicilina-ácido clavulânico, ceftazidima, cefotaxima e aztreonam, de acordo com o Clinical and Laboratory Standards Institute (CLSI) (CLSI, 2018).
Detecção de genes de resistência
O DNA utilizado nas análises moleculares foi obtido a partir de suspensões bacterianas constituídas por colônias frescas, que foram previamente cultivadas por 16-18 horas em ágar Luria Bertani e inoculadas em aproximadamente 300 μL de água ultrapura livre de nuclease homogeneizada com o auxílio de um agitador de tubo de vórtice (Vision Scientific).
A investigação de genes para β-lactâmicos e resistência às quinolonas foi realizada por PCR. As condições de ciclagem térmica foram as descritas pelos autores listados na tabela de primer.
Tabela 2: Primers utilizados para detectar os genes β-lactamases
| Cartilha | Sequências de 5′ 3′ | Gene alvo | Produto | Referências | |
| KPC | F | TGTCACTGTATCGCCGTC | blaKPC | | 1011pb | (Bratu et al., 2005) |
| R | CTCAGTGCTCTACAGAAAACC | ||||
| CTX-M | F | SCSATGTGCAGYACCAGTAA | blaCTXM | | 500pb | (Cao et al., 2002) |
| R | CCGCRATATGRTTGGTGGTG | ||||
| TEM | F | TCGGGGAAATGTGCGCG | blaTEM | | 700pb | |
| R | TGCTTAATCAGTGAGGCACC | ||||
| SHV | F | TTATCTCCCTGTTAGCCACC | blaSHV | | 900pb | |
| R | GATTTGCTGATTTCGCTCGG | ||||
| R | AAGCAGACTTGACCTGA | ||||
| qnrA | F | AGA GGA TTT CTC ACG CCA GG | qnrA | 580pb | (Cattoir et al., 2007). |
| R | TGC CAG GCA CAG ATC TTG AC | ||||
| qnrB | F | GGM ATH GAA ATT CGC CAC T | qnrB | 264pb | |
| R | TTT GCY GYY CGC CAG TCG AA | ||||
| qnrC | F | GGG TTG TAC ATT TAT TGA ATC | qnrC | 447pb | (Wang et al., 2009) |
| R | TCC ACT TTA CGA GGT TCT | ||||
| qnrD | F | CGA GAT CAA TTT ACG GGG AAT A | qnrD | 582pb | (Cavaco et al., 2009). |
Fonte: Autores.
Análise do perfil genético dos isolados
A comparação e identificação das variações genéticas no conteúdo de cepas bacterianas foi realizada por ERIC-PCR. Os primers utilizados para a reação foram ERIC-1R (5′ ATGTAAGCTCCTGGGGATTCAC-3′) e ERIC2 (5’AAGTAAGTGACTGGGGGGGCCG-3′) (Sigma Aldrich) de acordo com (VERSALOVIC et al., 1991), com modificação descrita por (FENDRI et al., 2013).
Capacidade de produção de biofilme
A capacidade de formação de biofilme foi avaliada de acordo com a metodologia proposta por (STEPANOVIĆ et al., 2000; STEPANOVIĆ et al., 2007). Cada cepa foi diluída em 108UFC/mL (0,5 na escala MacFarland) utilizando Caldo Trypticase de Soja (TSB) (Merck), e 200μL foram cultivados em três poços da microplaca de poliestireno de fundo plano de 96 poços (Nest®). Um total de 69 poços foram utilizados para testar 21 cepas; os outros 3 poços receberam o Salmonella Typhimurim ATCC 14028 como controle positivo, e 3 poços receberam o controle negativo (meio de cultura não inoculado). As placas contendo cepas de Salmonella spp. e controles foram incubadas a 35ºC por 96 horas. A placa foi lavada três vezes com solução salina tamponada com fosfato (PBS pH 7,2) e corada com violeta cristal a 1% por 15 minutos. Após lavagem três vezes com água destilada e secagem à temperatura ambiente, a absorbância foi lida em um leitor de microplacas Polaris (Celer®) a 492 nm de comprimento de onda (SERENO et al., 2017).
A densidade óptica (OD)[11] de cada cepa de Salmonella spp. foi obtida pela média aritmética da absorvância de três poços e esse valor foi comparado com a absorvância média dos controles negativos (ODnc). Em seguida, as cepas foram classificadas em nenhum produtor de biofilme (OD≤ODnc); produtor de biofilme fraco (ODnc<ODs≤2.ODnc); produtor moderado de biofilme (2.ODnc<ODs≤4.ODnc); e forte produção de biofilme (4.ODnc<ODs) classificação dada de acordo com (STEPANOVIĆ et al., 200; STEPANOVIĆ et al., 2007).
RESULTADOS
Prevalência de Salmonella spp. em amostras de carcaça de carne de aves
Das 61 amostras de carcaças de carne de aves coletadas nos mercados públicos, 34% (21) estavam contaminadas por Salmonella spp. A prevalência de Salmonella spp. em cada mercado público é expressa na figura 3, e os dados são detalhados na tabela 3.
Figura 2: Prevalência de isolados de Salmonella spp. de cada mercado público da cidade de Recife – Pernambuco, Brasil
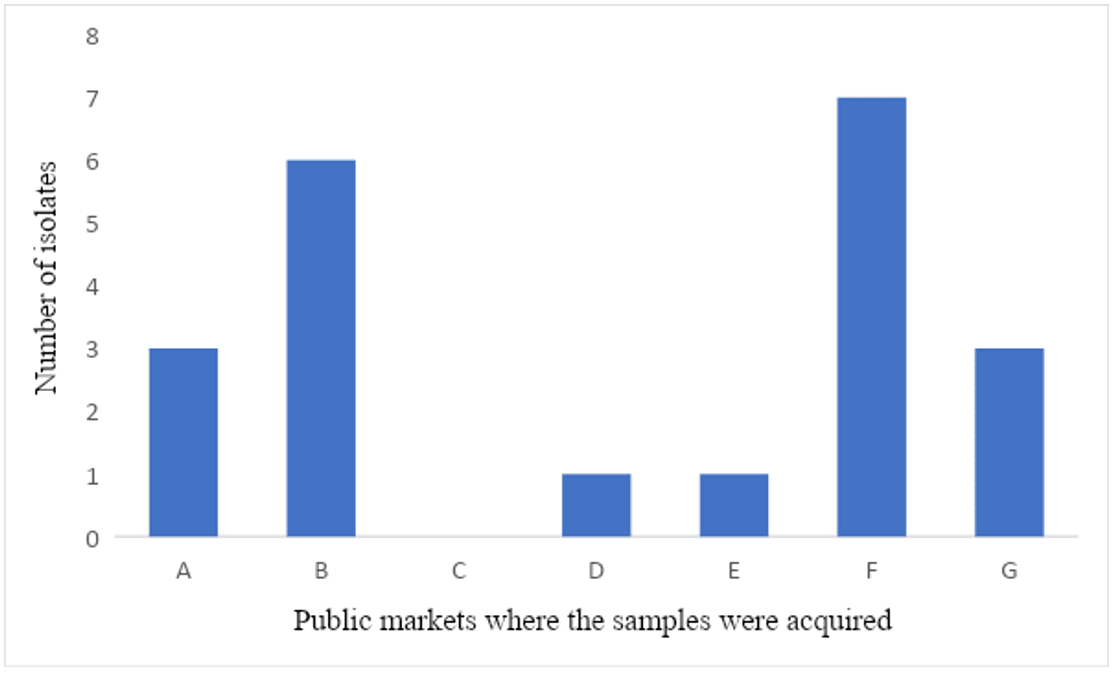
Tabela 3: Isolados de Salmonella spp. de cada mercado público da cidade do Recife – Pernambuco, Brasil
| Isolados | Mercado público | Ano |
| 1 | Um | 2018 |
| 2 | 2019 | |
| 3 | 2019 | |
| 4 | B | 2018 |
| 5 | 2018 | |
| 6 | 2018 | |
| 7 | 2018 | |
| 8 | 2018 | |
| 9 | 2018 | |
| 10 | D | 2019 |
| 11 | E | 2019 |
| 12 | F | 2019 |
| 13 | 2019 | |
| 14 | 2019 | |
| 15 | 2019 | |
| 16 | 2019 | |
| 17 | 2019 | |
| 18 | 2019 | |
| 19 | G | 2018 |
| 20 | 2019 | |
| 21 | 2019 |
Fonte: Autores.
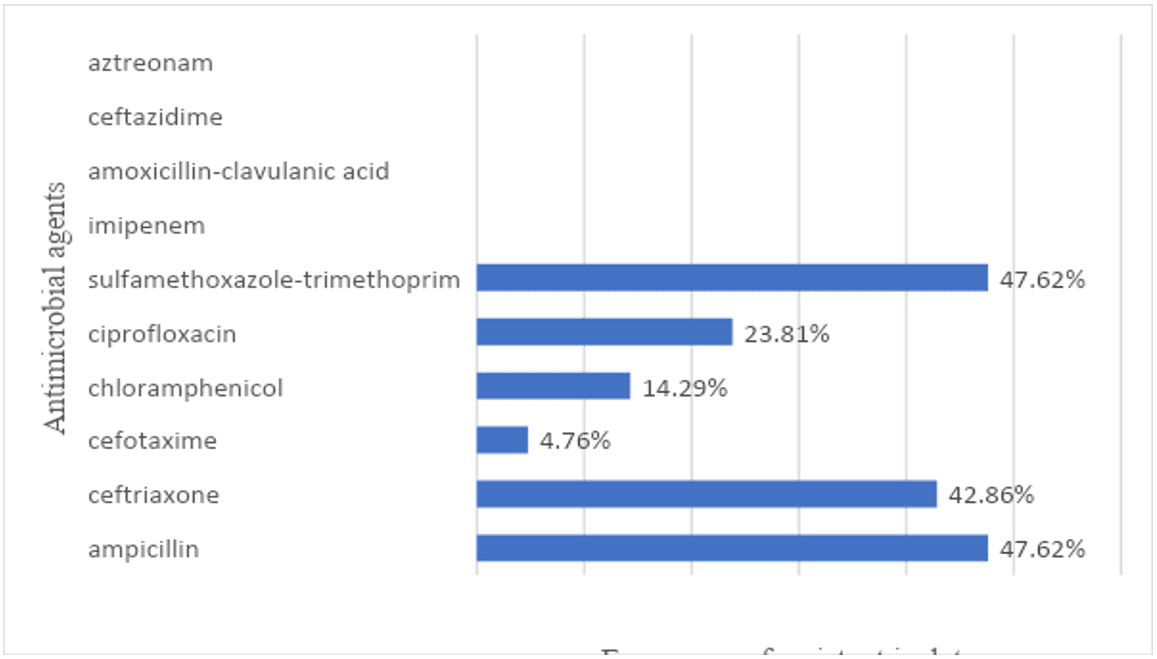
Figura 3: Resistência antimicrobiana a agentes antimicrobianos individuais em isolados de Salmonella spp. de carcaças de aves de capoeira comercializadas em mercados públicos da cidade de Recife – Brasil
Perfil de suscetibilidade
Dos 21 isolados, 13 (61,9%) foram resistentes a pelo menos um agente antimicrobiano. A resistência antimicrobiana a agentes antimicrobianos individuais entre isolados de Salmonella é expressa na figura.3. Os dados pormenorizados do teste de suscetibilidade antimicrobiana são expressos no quadro 4.
Tabela 4. Perfil de suscetibilidade antimicrobiana de isolados de Salmonella spp. de carcaças de frango adquiridas em mercados públicos da cidade do Recife – PE, entre 2018 e 2019
| Isolar | AMP | AMC | CRO | CTX | CAZ | IPM | Caixa automático | CLO | CIP | SUT |
| 1 | R | S | R | S | S | S | S | S | R | R |
| 2 | S | S | S | S | S | S | S | S | Eu | S |
| 3 | S | S | S | S | S | S | S | S | R | R |
| 4 | R | S | Eu | S | S | S | S | S | S | S |
| 5 | S | S | S | S | S | S | S | S | Eu | S |
| 6 | S | S | Eu | S | S | S | S | S | R | S |
| 7 | S | S | Eu | S | S | S | S | S | Eu | S |
| 8 | S | S | S | S | S | S | S | S | R | S |
| 9 | S | S | Eu | S | S | S | S | S | Eu | S |
| 10 | S | S | S | S | S | S | S | S | Eu | S |
| 11 | S | S | S | S | S | S | S | S | Eu | S |
| 12 | R | S | R | S | S | S | S | S | Eu | R |
| 13 | R | S | R | S | S | S | S | S | Eu | R |
| 14 | S | S | S | S | S | S | S | S | Eu | S |
| 15 | R | S | R | S | S | S | S | R | S | R |
| 16 | R | S | R | S | S | S | S | R | S | R |
| 17 | R | S | R | S | S | S | S | R | S | R |
| 18 | R | S | R | S | S | S | S | S | S | R |
| 19 | S | S | S | S | S | S | S | S | S | S |
| 20 | R | S | R | S | S | S | S | S | S | R |
| 21 | R | S | R | R | S | S | S | S | R | R |
AMP – Ampicilina, CLO – Cloranfenicol, CIP – Ciprofloxacina, CRO- Ceftriaxona, SUT – Sulfametoxazol-trimetoprim, IPM – Imipenem, AMC – amoxicilina-ácido clavulânico, CAZ – Ceftazidima, CTX- Cefotaxima e ATM – Aztreonam, S – Sensível; R- Resistente; Eu-. Intermediário. Fonte: Autores.
Pelo menos dois genes de resistência foram identificados em todos os isolados, dentre os quais a presença de genes de resistência que codificam β-lactamases (bla) e genes de resistência que codificam quinolonas (qnr) foram observados nos testes moleculares realizados no presente estudo, e os resultados estão detalhados na tabela 3.
Tabela 5: Genes de resistência detectados em cada isolado de Salmonella spp, recuperados de carcaças de aves adquiridas em mercados públicos da cidade de Recife – Pernambuco entre os anos de 2018 e 2019
| β-lactâmicos resistentes | Resistentes às quinolonas | |||||||
| Isolados | BlaTEM-like | BlaSHV-like | BlaCTX-M-like | BlaKPC-2 | qnrA | qnrB | qnrC | qnrD |
| 1 | + | – | – | – | + | + | – | – |
| 2 | + | – | – | – | + | + | – | – |
| 3 | + | – | – | – | + | + | – | + |
| 4 | + | – | – | – | + | + | – | – |
| 5 | + | – | – | – | + | + | – | + |
| 6 | + | – | – | – | + | + | – | + |
| 7 | + | – | – | – | + | + | – | + |
| 8 | + | – | – | – | + | + | – | + |
| 9 | + | – | – | – | + | + | – | + |
| 10 | + | – | + | – | – | + | – | – |
| 11 | + | – | – | – | – | + | – | – |
| 12 | + | – | + | – | – | + | – | – |
| 13 | + | – | + | – | – | + | – | – |
| 14 | + | – | + | – | – | + | – | – |
| 15 | + | – | + | – | – | + | – | – |
| 16 | + | – | + | – | – | + | – | + |
| 17 | – | – | + | – | – | + | – | + |
| 18 | + | – | + | – | – | + | – | + |
| 19 | + | – | – | – | – | + | – | + |
| 20 | + | – | – | – | – | + | – | + |
| 21 | + | – | – | – | + | + | – | + |
Fonte: Autores.
Relação clonal
Os isolados apresentaram um padrão variando de 3 a 9 tiras, o que revelou a presença de diferentes genótipos padrão indistinguíveis entre si e outros cujo padrão é diferente para apenas uma ou duas bandas. Alguns isolados de Salmonella spp. apresentaram padrões genéticos idênticos (2, 3, 4, 5, 6 e 7). As amostras 9, 11, 19, 20 e 21 foram intimamente relacionadas geneticamente, pois têm um padrão semelhante de tiras, diferindo apenas em uma (9, 21) ou duas tiras (11 e 20).
Formadores de biofilme
Todos os 21 isolados de Salmonella spp. testados eram produtores de biofilme, e esses isolados foram classificados como produtores de biofilme fracos de acordo com (STEPANOVIĆ et al., 2000; STEPANOVIĆ et al., 2007).
DISCUSSÃO
Este trabalho identificou Salmonella spp. contaminando carcaças de frango comercializadas em mercados públicos de uma metrópole brasileira. Além disso, detectamos os mecanismos de resistência presentes nesses isolados bacterianos e confirmamos que todos eles eram produtores de biofilme. Os resultados aqui apresentados mostraram vários isolados com proximidade genética e revelaram que os demais são clones.
Os isolados 2, 3, 4 e 5 têm um perfil genético idêntico, embora tenham sido coletados em dois mercados diferentes, Mercado A e Mercado B, que estão a cerca de 5 km de distância um do outro. Este achado indica que os vendedores podem ter o mesmo fornecedor, onde a contaminação por Salmonella MDR começou. O mercado F apresentou maior número de cepas caracterizadas como clones (12, 14, 15, 16 e 18). Além da possibilidade de um mesmo fornecedor, de acordo com um conjunto de falhas em condições higiênicas, como a exposição de produtos a poeira e insetos e o compartilhamento inadequado de utensílios como facas e recipientes entre os comerciantes, eles podem contribuir para a contaminação das carcaças, o que resulta na disseminação de isolados isogênicos (AZEVEDO, 2014).
A presença de Salmonella spp. na carne de aves de capoeira também foi observada nos estudos realizados por (BAPTISTA et al., 2018; MELO et al., 2020; YIN et al., 2021). No entanto, nossos achados diferem desses estudos devido à maior prevalência de isolados de Salmonella spp.
A contaminação por Salmonella spp. detectada nas amostras pode ter sido decorrente de contaminação prévia no próprio matadouro, devido a falhas no processo de abate (LOPES et al., 2007; VON RÜCKERT et al., 2009). Observou-se também que os comerciantes retiravam as carcaças da embalagem original e as colocavam em tigelas plásticas e posteriormente as vendiam à temperatura ambiente (88ºF), o que é considerado uma fraude. Essa temperatura promove a multiplicação de Salmonella spp., pois esses microrganismos podem se multiplicar em temperaturas em torno de 5°C a 45°C (MAHMOUD, 2012) Esse problema relacionado à temperatura também foi notado nos estudos realizados por (KHAN et al., 2018; JARQUIN et al., 2015).
Portanto, essa prática aumenta o risco de contaminação do produto, principalmente devido a práticas inadequadas de manuseio e condições higiênicas sanitárias inadequadas e ao que é observado nesses mercados públicos, pois verificou-se que mesas e bancos não foram limpos em 59% (36/61) dos locais. Em relação aos manipuladores, observou-se que 100% não tinham as unhas bem cortadas e limpas. Além disso, o mesmo funcionário que cuidava da carne era o mesmo no caixa. Consequentemente, essas falhas nos processos de boas práticas de manuseio observadas nesses mercados podem contribuir para a contaminação e disseminação de Salmonella spp. neste produto. Essa transferência pode ocorrer entre várias interações entre o homem, a comida e o meio ambiente. Portanto, a contaminação cruzada é a base para a disseminação de Salmonella spp.
A partir dos resultados do teste de suscetibilidade contra agentes antimicrobianos do presente estudo, observou-se que 38% (21/08) das cepas de Salmonella spp eram Resistentes a Múltiplas Drogas (MDR)[12], ou seja, bactérias resistentes a pelo menos três classes de agentes antimicrobianos (MAGIORAKOS et al., 2011).
A ocorrência de cepas de Salmonella spp resistentes a múltiplos antimicrobianos em carne de aves no Brasil é uma realidade crescente, devido aos dados relatados no presente estudo, e outras pesquisas realizadas por (ZIECH, 2015; BAPTISTA et al., 2018). Isso pode acontecer devido ao uso intensivo de agentes antimicrobianos na produção animal para tratar os animais e também para melhorar a produção.
A maior implicação em relação à disseminação de cepas zoonóticas multirresistentes está relacionada a falhas terapêuticas diante de diversas condições, tanto em casos humanos quanto animais. Além disso, essa realidade leva a perdas nos rebanhos de animais, uma vez que, quando presentes, essas cepas resistentes são difíceis de eliminar. A permanência de cepas patogênicas e multirresistentes na produção avícola é ainda mais preocupante, uma vez que a densidade animal é maior e, consequentemente, há uma maior disseminação dos microrganismos em um menor período de tempo.
Observou-se que apenas 4,28% (21/03) dos isolados foram resistentes ao cloranfenicol. Esse resultado pode refletir a proibição do uso desse agente como agente terapêutico e promotor de crescimento na produção animal no Brasil (PACHECO-SILVA et al., 2014).
Por outro lado, 42,85% (21/09) dos isolados eram resistentes à ceftriaxona, cefalosporina de 3ª geração. O uso dessa droga na produção animal é proibido no Brasil (MION et al., 2014). Portanto, esse resultado indica que esse ou outros fármacos da mesma classe antimicrobiana ainda estão sendo utilizados na produção animal no Brasil.
Além disso, 42,85 (9/21) dos isolados foram resistentes a três ou mais antimicrobianos. Então, ao comparar o perfil de resistência com os mercados, percebeu-se que os isolados de número 13, 15, 16, 17, 18, que eram resistentes a até três medicamentos, eram do mesmo mercado público (mercado público F). Sugere que pode ter havido contaminação cruzada no mercado, ou que essas carcaças de frango podem ter sofrido contaminação anterior no matadouro de aves.
Assim, a ocorrência de cepas de Salmonella MDR em alimentos de origem animal está aumentando no Brasil (BAPTISTA et al., 2018; MELO et al., 2020; VOSS-RECH et al., 2015). As infecções relacionadas a cepas multirresistentes estão associadas a alta morbidade e mortalidade, quando comparadas às sensíveis, uma vez que esses microrganismos representam uma barreira no tratamento de doenças humanas e animais.
A presença de tais genes de resistência detectados em isolados de Salmonella spp. de alimentos de origem animal representa uma séria ameaça à saúde pública, uma vez que a transmissão horizontal de genes de resistência ocorre principalmente por plasmídeos que codificam β-lactamases (GYLES, 2008). Além disso, esses genes são considerados de origem comunitária e são comuns em ambientes hospitalares (SUN et al., 2013). Assim, a disseminação de cepas resistentes aponta para a importância do controle de Salmonella spp na carne de aves de capoeira (BORGES et al., 2019; SIVASANKAR et al., 2020).
A presença de cepas formadoras de biofilme de Salmonella spp. observada no presente estudo aponta o risco que esse achado pode trazer para a indústria alimentícia e para a saúde pública. A capacidade de formar biofilme protegeu os microrganismos no interior do biofilme e, portanto, menos suscetíveis a fatores externos, como a ação de agentes antimicrobianos (ZIECH, 2015; SERENO et al., 2017; BORGES et al., 2018).
Além disso, os biofilmes promovem a permanência de microrganismos patogênicos e deteriorados, que causam doenças aos consumidores, e a depreciação do produto final devido a alterações físico-químicas e sensoriais (KASNOWSKY et al., 2010; SINGH et al., 2017).
Finalmente, esta investigação mostrou que a identificação de Salmonella spp. produtora de biofilme e portadora de diferentes genes β-lactamase e determinantes de resistência às quinolonas demonstra a capacidade dessas bactérias de acumular vários mecanismos de virulência e resistência a antimicrobianos. A pressão seletiva exercida pelo uso indiscriminado de antibióticos na agricultura é um fator importante para a aquisição de diferentes mecanismos de resistência e disseminação dessas cepas. Além disso, a disseminação de diferentes grupos clonais de Salmonella spp. MDR, em carcaças de frango, demonstrada neste estudo, atesta a necessidade de controles efetivos para conter esse microrganismo, que além de ser um risco para a saúde pública, também é responsável por perdas econômicas consideráveis.
APOIO FINANCEIRO
Os autores agradecem à Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) pela bolsa concedida.
REFERÊNCIAS
ALEKSHUN, M. N.; LEVY, S. B. Molecular Mechanisms of Antibacterial Multidrug Resistance. Cell, v. 128, n. 6, p. 1037–1050, 23 mar. 2007. Available from: https://doi.org/10.1016/j.cell.2007.03.004. Acessed in: 02 mar. 2021.
AZEVEDO, S. M. M. Farmacologia dos antibióticos beta-lactâmicos. Porto, Portugal: Universidade Fernando Pessoa, 2014. Available from: https://bdigital.ufp.pt/handle/10284/4412. Acessed in: 02 mar. 2021.
BAPTISTA, D. Q. et al. Prevalência e susceptibilidade antimicrobiana de sorotipos de Salmonella spp. isolados de frangos vivos e carcaças no estado do Rio de Janeiro. Pesquisa Veterinária Brasileira, v. 38, n. 7, p. 1278–1285, jul. 2018. Available from: https://doi.org/10.1590/1678-5150-PVB-5289. Acessed in: 02 mar. 2021.
BORGES, K. A. et al. Biofilm formation capacity of Salmonella serotypes at different temperature conditions. Pesquisa Veterinária Brasileira, v. 38, n. 1, p. 71–76, jan. 2018. Available from: https://doi.org/10.1590/1678-5150-pvb-4928. Acessed in: 02 mar. 2021.
BORGES, K. A.; MARTELO, E. B.; DOS SANTOS, L. A.; FURIAN, T. Q.; CISCO, I. C.; MANTO, L.; DOS SANTOS, L. R. Detection and quantification of Salmonella spp. in poultry slaughterhouses of southern Brazil. The Journal of Infection in Developing Countries, [S. l.], v. 13, n. 05, p. 455–460, 2019. DOI: 10.3855/jidc.11107. Available from: https://jidc.org/index.php/journal/article/view/32053516. Acessed in: 02 mar. 2021.
BRATU, S.; LANDMAN, D.; ALAM, M.; TOLENTINO, E.; QUALE, J. Detection of KPC Carbapenem-Hydrolyzing Enzymes in Enterobacter spp. from Brooklyn, New York. Antimicrobial Agents and Chemotherapy, v. 49, n. 2, p. 776–778, fev. 2005. Available from: https://doi.org/10.1128/AAC.49.2.776-778.2005. Acessed in: 02 mar. 2021.
BRASIL. Manual Integrado de Vigilância, Prevenção e Controle de Doenças Transmitidas por Alimentos. Ministério da Saúde, Secretária de Vigilância em Saúde, 2019. Available from: http://portalarquivos2.saude.gov.br/images/pdf/2019/fevereiro/15/Apresenta oSurtos-DTA—Fevereiro-2019.pdf. Acessed in: 13 Aug. 2019.
BONI, H. F. K.; CARRIJO, A. S.; FASCINA, V. B. Detection of Salmonella spp. in broiler buildings and stuff of slaughterhouse in the central region of Mato Grosso do Sul. R. bras. Saúde Prod. Anim., 2011. Available from: http://revistas.ufba.br/index.php/rbspa/article/view/1949. Acessed in: 02 mar. 2021.
CAO, V.; LAMBERT, T.; NHU, D. Q.; LOAN, H. K.; HOANG, N. K.; ARLET, G.; COURVALIN, P. Distribution of Extended-Spectrum β-Lactamases in Clinical Isolates of Enterobacteriaceae in Vietnam. Antimicrobial Agents and Chemotherapy, v. 46, n. 12, p. 3739–3743, dez. 2002. Available from: https://doi.org/10.1128/AAC.46.12.3739-3743.2002. Acessed in: 02 mar. 2021.
CATTOIR, V.; POIREL, L.; ROTIMI, V.; SOUSSY, C. J.; NORDMANN, P. Multiplex PCR for detection of plasmid-mediated quinolone resistance qnr genes in ESBL-producing enterobacterial isolates. Journal of Antimicrobial Chemotherapy, v. 60, n. 2, p. 394–397, 1 ago. 2007. Available from: https://doi.org/10.1093/jac/dkm204. Acessed in: 02 mar. 2021.
CAVACO, L. M.; HASMAN, H.; XIA, S.; AARESTRUP, F. M. qnrD, a novel gene conferring transferable quinolone resistance in Salmonella enterica serovar Kentucky and Bovismorbificans strains of human origin. Antimicrobial Agents and Chemotherapy, v. 53, n. 2, p. 603–608, fev. 2009. Available from: https://doi.org/10.1128/AAC.00997-08. Acessed in: 02 mar. 2021.
CENTRES FOR DISEASES, CONTROL AND PREVENTION – CDC. Salmonella. Centres For Diseases, Control And Prevention, 2020. Available from: https://www.cdc.gov/salmonella/index.html. Acessed in: 02 mar. 2021.
DAVEY, M. E.; O’TOOLE, G. A. Microbial Biofilms: from Ecology to Molecular Genetics. Microbiology and Molecular Biology Reviews, v. 64, n. 4, p. 847–867, dez. 2000. Available from: https://doi.org/10.1128/mmbr.64.4.847-867.2000. Acessed in: 02 mar. 2021.
DUARTE, D. A. M. et al. Occurrence of Salmonella spp. in broiler chicken carcasses and their susceptibility to antimicrobial agents. Brazilian Journal of Microbiology, v. 40, n. 3, p. 569–573, set. 2009. Available from: https://doi.org/10.1590/S1517-83822009000300020. Acessed in: 02 mar. 2021.
FENDRI, I.; BEN HASSENA, A.; GROSSET, N.; BARKALLAH, M.; KHANNOUS, L.; CHUAT, V.; GAUTIER, M.; GDOURA, R. Genetic Diversity of Food-Isolated Salmonella Strains through Pulsed Field Gel Electrophoresis (PFGE) and Enterobacterial Repetitive Intergenic Consensus (ERIC-PCR). PLOS ONE, v. 8, n. 12, p. e81315, 3 dez. 2013. Available from: https://doi.org/10.1371/journal.pone.0081315. Acessed in: 02 mar. 2021.
FITCH, F. M.; CARMO-RODRIGUES, M. S.; OLIVEIRA, V. G. S.; GASPARI, M. V.; DOS SANTOS, A.; DE FREITAS, J. B.; PIGNATARI, A. C. C. β-Lactam Resistance Genes: Characterization, Epidemiology, and First Detection of blaCTX-M-1 and blaCTX-M-14 in Salmonella spp. Isolated from Poultry in Brazil—Brazil Ministry of Agriculture’s Pathogen Reduction Program. Microbial Drug Resistance, v. 22, n. 2, p. 164–171, 8 mar. 2016. Available from: https://doi.org/10.1089/mdr.2015.0143. Acessed in: 02 mar. 2021.
FLEMMING, H. C.; WINGENDER, J.; SZEWZYK, U.; STEINBERG, P.; RICE, S. A.; KJELLEBERG, S. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology, v. 14, n. 9, p. 563–575, 11 ago. 2016. Available from: https://doi.org/10.1038/nrmicro.2016.94. Acessed in: 02 mar. 2021.
GRIMONT, P.; WEILL, F. X. Antigenic formulae of the Salmonella servovars: WHO Collaborating Centre for Reference and Research on Salmonella. 9th Ed. Inst. Pasteur, 2007. 1–166 p.
GYLES, C. L. Antimicrobial resistance in selected bacteria from poultry. Animal Health Research Reviews, v. 9, n. 2, p. 149–158, 2008. Available from: https://doi.org/10.1017/S14662523080015522. Acessed in: 02 mar. 2021.
JARQUIN, C.; ALVAREZ, D.; MORALES, O.; MORALES, A. J.; LÓPEZ, B.; DONADO, P.; VALENCIA, M. F.; ARÉVALO, A.; MUÑOZ, F.; WALLS, I.; DOYLE, M. P.; ALALI, W. Q. Salmonella on Raw Poultry in Retail Markets in Guatemala: Levels, Antibiotic Susceptibility, and Serovar Distribution. Journal of Food Protection, v. 78, n. 9, p. 1642–1650, set. 2015. Available from: https://doi.org/10.4315/0362-028X.JFP-15-117. Acessed in: 02 mar. 2021.
KASNOWSKY, M. C. et al. Formação de biofilme na indústria de alimentos e métodos de validação de superfícies. Revista Científica Eletrônica de Medicina Veterinária, v. 15, p. 1679–7353, jul. 2010. Available from: http://faef.revista.inf.br/imagens_arquivos/arquivos_destaque/fxPTiYWerLkT9Si_2013-6-25-16-32-0.pdf. Acessed in: 02 mar. 2021.
KHAN, A. S.; GEORGES, K.; RAHAMAN, S.; ABDELA, W.; ADESIYUN, A. A. Prevalence and serotypes of Salmonella spp. on chickens sold at retail outlets in Trinidad. PLOS ONE, v. 13, n. 8, p. e0202108, ago. 2018. Available from: https://doi.org/10.1371/journal.pone.0202108. Acessed in: 02 mar. 2021.
LOPES, M.; GALHARDO, J. A.; DE OLIVEIRA, T.; TAMANINI, R.; SANCHES, F.; MULLER, E. Pesquisa de Salmonella spp. e microrganismos indicadores em carcaças de frango e água de tanques de pré-resfriamento em abatedouro de aves. Semina: Ciências Agrárias, v. 28, n. 3, p. 465–476, 30 ago. 2007. Available from: https://doi.org/10.5433/1679-0359.2007v28n3p465. Acessed in: 02 mar. 2021.
MAGIORAKOS, A. P.; SRINIVASAN, A.; CAREY, R. B.; CARMELI, Y.; FALAGAS, M. E.; GISKE, C. G.; HARBARTH, S.; HINDLER, J. F.; KAHLMETER, G.; OLSSON-LILJEQUIST, B.; PATERSON, D. L.; RICE, L. B.; STELLING, J.; STRUELENS, M. J.; VATOPOULOS, A.; WEBER, J. T.; MONNET, D. L. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clinical microbiology and infection: the official publication of the European Society of Clinical Microbiology and Infectious Diseases, v. 18, n. 3, p. 268–281, 2012. Available from: https://pubmed.ncbi.nlm.nih.gov/21793988/. Acessed in: 02 mar. 2021.
MAHMOUD, B. S. M. Salmonella – A Dangerous Foodborne Pathogen, Nursing times, 2012. Available from: https://doi.org/10.1049/cm.1989.0039. Acessed in: 02 mar. 2021.
MELO, R. T. et al. Salmonella Minnesota de origem avícola apresenta fatores de virulência e risco potencial aos humanos. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v. 72, n. 4, p. 1353–1362, jul. 2020. Available from: https://doi.org/10.1590/1678-4162-10884. Acessed in: 02 mar. 2021.
MION, L.; COLLA, F. L.; CISCO, I. C.; WEBBER, B.; DIEDRICH, L. N.; PILOTTO, F.; RODRIGUES, L. B.; NASCIMENTO, V. P. do; SANTOS, L. R. dos. Perfil de resistência a antimicrobianos por Salmonella Heidelberg isoladas de abatedouro avícola em 2005 e 2009. Acta Scientiae Veterinariae, v. 42, n. 1, p. 1–5, 2014. Available from: https://www.redalyc.org/articulo.oa?id=289029240017. Acessed in: 02 mar. 2021.
PACHECO-SILVA, É.; SOUZA, J. R. de; CALDAS, E. D. Resíduos de medicamentos veterinários em leite e ovos. Química Nova, v. 37, n. 1, p. 111–122, 2014. Available from: https://doi.org/10.1590/S0100-40422014000100020. Acessed in: 02 mar. 2021.
PENG, M.; SALAHEEN, S.; BISWAS, D. Animal Health: Global Antibiotic Issues. Encyclopedia of Agriculture and Food Systems, p. 346–357, jan. 2014. Available from: https://doi.org/10.1016/B978-0-444-52512-3.00187-X. Acessed in: 02 mar. 2021.
REZENDE, C. S. M. e; MESQUITA, A. J. de; ANDRADE, M. A.; LINHARES, G. F. C.; MESQUITA, A. Q. de; MINAFRA, C. S. Sorovares de Salmonella isolados de carcaças de frangos de corte abatidos no Estado de Goiás, Brasil, e perfil de resistência a antimicrobianos. Revista Portuguesa de Ciências Veterinárias, Lisboa, v. 100, n. 555-556, p. 199-203, jul./dez. 2005. Available from: http://repositorio.bc.ufg.br/handle/ri/12309. Acessed in: 02 mar. 2021.
RIBEIRO, A. R. et al. Salmonella spp. in raw br
oiler parts: occurrence, antimicrobial resistance profile and phage typing of the Salmonella Enteritidis isolates. Brazilian Journal of Microbiology, v. 38, n. 2, p. 296–299, abr. 2007. Available from: https://doi.org/10.1590/S1517-83822007000200021. Acessed in: 02 mar. 2021.
SERENO, M. et al. Antimicrobial Susceptibility and Biofilm Production by Salmonella sp. Strains Isolated from Frozen Poultry Carcasses. Brazilian Journal of Poultry Science, v. 19, n. 1, p. 103–108, jan. 2017. Available from: https://doi.org/10.1590/1806-9061-2016-0268. Acessed in: 02 mar. 2021.
SINGH, S.; SINGH, S. K.; CHOWDHURY, I.; SINGH, R. Understanding the Mechanism of Bacterial Biofilms Resistance to Antimicrobial Agents. The Open Microbiology Journal, v. 11, n. 1, p. 53–62, mai. 2017. Available from: https://doi.org/10.2174/1874285801711010053. Acessed in: 02 mar. 2021.
SIVASANKAR, C.; JHA, N. K.; GHOSH, R.; SHETTY, P. H. Anti quorum sensing and anti virulence activity of tannic acid and it’s potential to breach resistance in Salmonella enterica Typhi / Paratyphi A clinical isolates. Microbial Pathogenesis, v. 138, p. 103813, 1 jan. 2020. Available from: https://doi.org/10.1016/j.micpath.2019.103813. Acessed in: 02 mar. 2021.
STEPANOVIĆ, S.; VUKOVIĆ, D.; DAKIĆ, I.; SAVIĆ, B.; ŠVABIĆ-VLAHOVIĆ, M. A modified microtiter-plate test for quantification of staphylococcal biofilm formation. Journal of Microbiological Methods, v. 40, n. 2, p. 175–179, 1 abr. 2000. Available from: https://doi.org/10.1016/S0167-7012(00)00122-6. Acessed in: 02 mar. 2021.
STEPANOVIĆ, S.; VUKOVIĆ, D.; HOLA, V.; DI BONAVENTURA, G.; DJUKIĆ, S.; ĆIRKOVIĆ, I.; RUZICKA, F. Quantification of biofilm in microtiter plates: overview of testing conditions and practical recommendations for assessment of biofilm production by staphylococci. APMIS: acta pathologica, microbiologica, et immunologica Scandinavica, v. 115, n. 8, p. 891–899, 2007. Available from: https://pubmed.ncbi.nlm.nih.gov/17696944/. Acessed in: 02 mar. 2021.
SUN, D. D.; MA, X. X.; HU, J.; TIAN, Y.; PANG, L.; SHANG, H.; CUI, L. Z. Epidemiological and molecular characterization of community and hospital acquired Staphylococcus aureus strains prevailing in Shenyang, Northeastern China. The Brazilian Journal of Infectious Diseases, v. 17, n. 6, p. 682–690, 1 nov. 2013. Available from: https://doi.org/10.1016/j.bjid.2013.02.007. Acessed in: 02 mar. 2021.
THRELFALL, E. J. Antimicrobial drug resistance in Salmonella: problems and perspectives in food- and water-borne infections. FEMS Microbiology Reviews, v. 26, n. 2, p. 141–148, 1 jun. 2002. Available from: https://academic.oup.com/femsre/article/26/2/141/653178. Acessed in: 02 mar. 2021.
VERSALOVIC, J.; KOEUTH, T.; LUPSKI, R. Distribution of repetitive DNA sequences in eubacteria and application to finerpriting of bacterial enomes. Nucleic Acids Research, v. 19, n. 24, p. 6823–6831, dez. 1991. Available from: https://doi.org/10.1093/nar/19.24.6823. Acessed in: 02 mar. 2021.
VOSS-RECH, D.; VAZ, C. S. L.; ALVES, L.; COLDEBELLA, A.; LEAO, J. A.; RODRIGUES, D. P.; BACK, A. A temporal study of Salmonella enterica serotypes from broiler farms in Brazil. Poultry Science, v. 94, n. 3, p. 433–441, 1 mar. 2015. Available from: https://doi.org/10.3382/ps/peu081. Acessed in: 02 mar. 2021.
VON RÜCKERT, D. A. S. et al. Pontos críticos de controle de Salmonella spp. no abate de frangos. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v. 61, n. Arq. Bras. Med. Vet. Zootec., vol. 61, n. 2, p. 326–330, abr. 2009. Available from: https://doi.org/10.1590/S0102-09352009000200007. Acessed in: 02 mar. 2021.
WANG, M.; GUO, Q.; XU, X.; WANG, X.; YE, X.; WU, S.; HOOPER, D. C.; WANG, M. New plasmid-mediated quinolone resistance gene, qnrC, found in a clinical isolate of Proteus mirabilis. Antimicrobial Agents and Chemotherapy, v. 53, n. 5, p. 1892–1897, maio 2009. Available from: https://doi.org/10.1128/AAC.01400-08. Acessed in: 02 mar. 2021.
YIN, X.; M’IKANATHA, N. M.; NYIRABAHIZI, E.; MCDERMOTT, P. F.; TATE, H. Antimicrobial resistance in non-Typhoidal Salmonella from retail poultry meat by antibiotic usage-related production claims – United States, 2008–2017. International Journal of Food Microbiology, v. 342, p. 109044, 16 mar. 2021. Available from: https://doi.org/10.1016/j.ijfoodmicro.2021.109044. Acessed in: 02 mar. 2021.
ZIECH, R. E. Caracterização de Salmonella sp. isolada de indústrias de aves baseada na formação de biofilmes, tolerância a sanitizantes e resistência a antimicrobianos. Dissertação (Mestrado em Ciência Animal) – Programa de Pós-Graduação em Ciência Animal, Universidade Federal do Paraná. Palotina, 2015.
APÊNDICE – NOTA DE RODAPÉ
11. Optical Density (OD).
12. Microbial Drug Resistance (MDR).
[1] Doutoranda do Programa de Pós-Graduação em Biociência Animal. ORCID: 0000-0003-0698-6125. CURRÍCULO LATTES: http://lattes.cnpq.br/5170743479462841.
[2] Doutoranda do Programa de Pós-Graduação em Biociência Animal. ORCID: 0000-0001-9410-7631. CURRÍCULO LATTES: http://lattes.cnpq.br/4532231119888940.
[3] Doutoramento em Genética. ORCID: 0000-0003-0698-6125. CURRÍCULO LATTES: http://lattes.cnpq.br/4891800920829895.
[4] Licenciatura em Medicina Veterinária. ORCID: 0000-0001-9273-5204. CURRÍCULO LATTES: http://lattes.cnpq.br/8329028352662293.
[5] M.Sc. em Biociência Animal. ORCID: 0000-0002-4913-8313. CURRÍCULO LATTES: http://lattes.cnpq.br/4967459162060058.
[6] Mestre em Biologia Celular e Molecular Aplicada. ORCID: 0000-0001-7764-0573. CURRÍCULO LATTES: http://lattes.cnpq.br/6281410502740086.
[7] Bacharel em Ciências Biológicas.ORCID: 0000-0001-7928-7435. CURRÍCULO LATTES: http://lattes.cnpq.br/9549169192404311.
[8] Doutor em Bioquímica e Fisiologia, Mestre em Fisiologia, Biólogo. ORCID: 0000-0003-1493-7964. CURRÍCULO LATTES: http://lattes.cnpq.br/9044747136928972.
[9] Doutor. ORCID: 0000-0002-1993-0350.
[10] Conselheiro. Doutor do Programa de Pós-Graduação em Biociência Animal. ORCID: 0000-0002-1289-2902. CURRÍCULO LATTES: http://lattes.cnpq.br/5998863169551704.
Enviado: 27 de março de 2023.
Aprovado: 08 de abril de 2023.