ARTÍCULO ORIGINAL
SILVA, Maria Goretti Varejão da [1], ANDRADE, Jéssica Martins de [2], MOURA, Fernanda Maria de Lino [3], MEDEIROS, Anna Karolyne de Araujo [4], CORDEIRO, Geovania de Souza [5], MELO, Nataly Sayonara da Silva [6], ROLIM, Maria Betânia Queiroz [7], MOURA, Vilton Edson Figueirôa de [8], SILVA, Daniel Dias da [9], SOARES, Anísio Francisco [10], MEDEIROS, Elizabeth Sampaio de [11]
SILVA, Maria Goretti Varejão da. et al. Enterococcus spp. resistente a antimicrobianos y formadores de biofilm en queso de coalho. Revista Científica Multidisciplinar Núcleo do Conhecimento. Año 08, Ed. 06, vol. 01, pp. 05-31. Junio de 2023. ISSN: 2448-0959, Enlace de acceso: https://www.nucleodoconhecimento.com.br/biologia-es/antimicrobianos-y-formadores, DOI: 10.32749/nucleodoconhecimento.com.br/biologia-es/antimicrobianos-y-formadores
RESUMEN
El objetivo de este estudio fue realizar una revisión de la literatura sobre Enterococcus spp. resistente a antimicrobianos y cepas formadoras de biofilme en queso de coalho originario del Noreste de Brasil. Se observó el impacto de la presencia de bacterias enterocócicas formadoras de biofilme en el queso de coalho y sus efectos en la salud pública de los consumidores de este tipo de alimento. Sin embargo, se necesitan más estudios microbiológicos sobre el queso de coalho debido a su importancia económica y de salud pública.
Palabras clave: Enterococcus spp, Microorganismos, Quesos, Resistencia a Medicamentos, Salud Pública.
INTRODUCCIÓN
El queso de coalho es un alimento típico brasileño presente en la región del Noreste durante más de 150 años y ampliamente consumido en esta región. Se elabora a partir de leche cruda o pasteurizada. Estados como Pernambuco, Ceará, Rio Grande do Norte y Paraíba son los principales productores de este tipo de queso. Su importancia económica es considerable y significativa para los productores de leche, especialmente aquellos que no tienen acceso a las unidades de procesamiento de leche (SILVA et al., 2012).
Los enterococos, bacterias Gram-positivas aisladas del suelo, aguas superficiales y del mar, también en asociación con plantas, están presentes en productos alimenticios fermentados. Son Bacterias Ácido Lácticas (BAL) ampliamente utilizadas como culturas iniciadoras en la industria láctea para la producción de queso, yogur y leche fermentada (GIRAFFA, 2003; GIRAFFA y ROSSETTI, 2004). Por lo general, no causan efectos adversos en individuos saludables, pero también pueden actuar como patógenos oportunistas relacionados con infecciones nosocomiales, especialmente en pacientes inmunocomprometidos (LOSSOUARN et al., 2019).
Las infecciones nosocomiales causadas por la microbiota intra-hospitalaria y en ocasiones condicionadas por la microbiota de los profesionales de la salud y del propio paciente son un problema de gran importancia clínica y epidemiológica. Debido a las altas tasas de morbilidad y mortalidad, así como a los largos días de hospitalización y al desperdicio de recursos económicos, se consideran una situación emergente en todo el mundo (MONTOYA et al., 2015).
Además, la resistencia intrínseca de Enterococcus spp. a varios antimicrobianos y la adquisición de resistencia a otros, como la vancomicina, que se utiliza con frecuencia en el tratamiento de pacientes con infecciones graves causadas por bacterias Gram-positivas, ha llevado a la aparición de E. faecalis como un patógeno hospitalario relevante (ANDRADE, 2018).
Enterococcus spp. se describe como la causa de al menos el 10% de las infecciones hospitalarias, según Hollenbeck y Rice (2012), y las principales especies asociadas a estas infecciones, E. faecalis (85-90%) y E. faecium, presentan resistencia intrínseca a varios antimicrobianos, como β-lactámicos (penicilina y cefalosporinas), aminoglicósidos (estreptomicina), lincosamidas (clindamicina en E. faecalis), estreptograminas (o combinación quinupristina-dalfopristina en E. faecalis) y sulfametoxazol-trimetoprima. El uso descontrolado de antimicrobianos ha provocado un aumento de cepas resistentes al tratamiento, y esta situación se vuelve más preocupante cuando los microorganismos se presentan en forma de biofilm. Las estructuras complejas de grupos microbianos protegidos por una capa de exopolisacáridos (EPS), los biofilms impiden la penetración de medicamentos y dificultan el tratamiento de las infecciones (COSTERTON et al., 1999; URQUHART et al., 2019).
A pesar de lo anterior, aún existe una falta de registros en la literatura sobre Enterococcus spp. y cepas formadoras de biofilm aisladas de alimentos de origen animal. Por lo tanto, investigar estos microorganismos con tales características en alimentos de origen animal se vuelve relevante.
METODOLOGÍA
El presente estudio describe una revisión de literatura cualitativa y narrativa, en la cual se seleccionaron finalmente 90 artículos para su inclusión y lectura completa. La base de datos utilizada incluyó artículos disponibles en Google Scholar, Scielo y PubMed, donde se seleccionaron 34, 14 y 42 artículos respectivamente para su posible inclusión final en la revisión. La selección de los artículos se llevó a cabo durante el año 2020 y se consideraron artículos publicados en español, inglés y portugués. Se seleccionaron artículos que abordaran el tema propuesto por la presente investigación. Durante la búsqueda de material bibliográfico, se utilizaron las palabras clave: enterococcus, enterococcus durans, enterococcus faecalis, enterococcus faecium, queso, queso de coalho, queso tipo coalho, resistencia, resistencia antimicrobiana, infección hospitalaria, infección nosocomial, biofilm, formación de biofilm.
La revisión se produjo mediante la lectura, análisis, interpretación y síntesis de la información obtenida en las obras seleccionadas que se consideraron relevantes para el tema abordado en el proyecto. Los criterios de exclusión del material bibliográfico fueron: trabajos publicados cuyos resultados no se aplicaran a los objetivos de esta revisión y artículos cuyo tema de estudio no estuviera relacionado con el tema. Los criterios de inclusión tuvieron en cuenta artículos que abordaran la especificidad del tema en cuestión y artículos y tesis publicados en los idiomas español, inglés y portugués. No hubo restricciones en cuanto al año de publicación de los estudios.
REVISIÓN DE LA LITERATURA
LA IMPORTANCIA DE LA LECHE Y EL QUESO EN BRASIL
Según datos del Instituto Brasileño de Geografía y Estadística (IBGE, 2018), la producción de leche en Brasil tiene un gran impacto a nivel mundial, siendo el quinto país con mayor producción en el mundo, después de la Unión Europea, Estados Unidos, India y China.
Los productos lácteos bajo inspección sanitaria en Brasil captaron 24,45 mil millones de litros de leche en 2018, un aumento del 0,5% con respecto a 2017 (IBGE, 2019), lo que es de gran importancia ya que la leche es un producto rico en nutrientes y energía, se considera un alimento completo y se recomienda su consumo para personas de todas las edades. Producida en todo el mundo, la leche se utiliza ampliamente en la alimentación infantil y es un alimento de primera línea en la lucha contra la mortalidad infantil en países en desarrollo (LUNA, 2012).
El queso es un producto que puede ser madurado o consumido fresco, obtenido mediante la separación parcial o total del suero de la leche, la leche reconstituida o el suero lácteo, que se coagulan con la ayuda de enzimas, cuajo, bacterias, ácidos orgánicos aislados o combinados (BRASIL, 1996).
Debido a sus propiedades sensoriales y nutricionales, el queso es un producto lácteo tradicional con una gran aceptación en el mercado, presentado para su consumo en diversas variedades que difieren en tipo, sabor, color, forma y aroma, buscando satisfacer los múltiples gustos de sus consumidores (NOGUEIRA, 2006).
Los quesos son alimentos de alto valor biológico, ricos en proteínas, calcio, fósforo, zinc, yodo, selenio, vitaminas y oligoelementos. En el mundo existen más de 1,000 tipos de quesos producidos a partir de diferentes tipos de leche y procesos de producción diferenciados (LÁCTEA BRASIL, 2006).
Con el desarrollo tecnológico, han surgido muchas variedades a nivel nacional, algunas de ellas de expresión regional, como el queso de coalho (BORGES et al., 2003). Según Andrade (2006), el nombre proviene del proceso de coagulación, observado por primera vez debido a la acción de enzimas coagulantes presentes en el estómago de animales herbívoros. Uno de los productos típicos de la región Nordeste, el queso de coalho, tiene un valor cultural y socioeconómico significativo, forma parte de la alimentación diaria y se consume comúnmente como aperitivo o acompañamiento de otros alimentos. Su producción se transmite de generación en generación, principalmente a través de métodos artesanales, utilizando conocimientos prácticos trad
icionales transmitidos en el entorno familiar (SOUZA et al., 2014). Cuando se elabora de forma artesanal, su producción sufre ciertas adaptaciones según la habilidad, estructura física y económica del fabricante. Como resultado, los quesos producidos tienen características organolépticas que les confieren una identidad regional y cultural propia de cada región (MENEZES, 2013).
El queso de coalho, tanto artesanal como industrializado, puede no presentar seguridad y estandarización microbiológica debido a que a menudo es manipulado y elaborado en condiciones higiénico-sanitarias insatisfactorias (ANDRADE, 2006), siendo considerado un alimento altamente susceptible a la contaminación (SANTANA et al., 2008).
Para regular su producción, existen algunas regulaciones, como el Reglamento Técnico de Identidad y Calidad de Productos Lácteos, que establece los estándares de identidad y los requisitos mínimos de calidad que el queso de coalho debe cumplir para ser destinado al consumo humano (FREITAS FILHO et al., 2009). La Ley Federal n.º 13.860 de 18/07/2019, sancionada por el Presidente de la República de Brasil, regula la producción y comercialización de queso artesanal y productos lácteos relacionados, mientras que la Portaria ADAGRO n.º 007 de 04/01/2018 aprueba el Reglamento Técnico de Identidad y Calidad del Queso de Coalho Artesanal en Pernambuco (Imprensa Nacional, 2019; DOE Pernambuco, 2018).
El queso industrializado (tipo A) sigue las normativas del MAPA, como la Instrucción Normativa n.º 5 de 14/02/2017, que establece los requisitos para la evaluación de la equivalencia con el Sistema Único de Atención Sanitaria Agropecuaria en cuanto a la estructura física, dependencias y equipamiento de las instalaciones agroindustriales de pequeña escala que producen productos de origen animal. Por último, la Portaria MAPA 368/97 establece el reglamento técnico sobre las condiciones higiénico-sanitarias y las Buenas Prácticas de Fabricación para establecimientos de procesamiento/industrialización de alimentos.
ENTEROCOCCUS SPP.
Las bacterias Enterococcus spp. son cocos Gram-positivos de forma ovalada que forman cadenas cortas y pertenecen a la familia Enterococcaceae, con un total de 58 especies descritas (PARTE, 2014). Estos microorganismos pueden crecer facultativamente en ambientes ricos en oxígeno o con baja concentración de oxígeno. Son microorganismos extremadamente resistentes a diferentes condiciones ambientales, tolerando temperaturas variables de 10°C a 45°C, así como ambientes hipotónicos con un contenido de hasta 6.5% de cloruro de sodio y ambientes hipertónicos con un pH que varía de ácido (pH entre 4 y 9.6), y pueden soportar hasta 60°C durante 30 minutos (KONEMAN et al., 2014).
El término “Enterococcus” fue descrito por primera vez por Thiercelin en 1899, cuando describió bacterias comensales con la capacidad de volverse patógenas. Debido a su morfología y algunas similitudes bioquímicas, los enterococos fueron inicialmente considerados como parte del género Streptococcus (SOLACHE, 2019). Son cocos anaeróbicos facultativos que forman cadenas de varias longitudes. Son habitantes comensales del tracto gastrointestinal de humanos y otros mamíferos, con la capacidad de sobrevivir en entornos como hospitales (ARIAS y MURRAY, 2012).
Enterococcus spp. y sus especies provienen de diversas fuentes, como el entorno, animales y humanos. Estos microorganismos son de gran importancia ya que forman parte de la microbiota normal de animales y humanos, con una distribución similar en ambos (FISCHER y PHILLIPS, 2009). Constituyen un grupo complejo de bacterias que desempeñan un doble papel en los alimentos, ya que algunas cepas se utilizan como cultivos iniciadores en quesos y alimentos enlatados, lo que confiere sabor, aroma y textura a estos productos, mientras que otras cepas están relacionadas con la degradación de alimentos como posibles reservorios de enterococos que albergan determinantes de virulencia y resistencia (TERRA et al., 2018).
A través de la contaminación fecal, la piel de los animales, el agua, los equipos de ordeño y los tanques de expansión, Enterococcus spp. puede contaminar la leche y sus derivados. Las bacterias Enterococcus se encuentran con frecuencia en la leche y sus derivados, especialmente en quesos, debido a su resistencia a la pasteurización por ser termorresistentes y a la refrigeración por ser psicrotróficas (PORTO et al., 2016).
Enterococcus spp. forma parte de la microbiota involucrada en varios procesos de fermentación de alimentos, como los que implican leche, carne y vegetales. Son componentes naturales de los alimentos y desempeñan un papel importante en la maduración del queso y en la mejora del sabor. Estos microorganismos tienen una amplia capacidad de adaptación para resistir condiciones adversas como temperatura, pH, hiperosmolaridad y desecación prolongada (ALI et al., 2014).
Su presencia en alimentos ha sido motivo de preocupación para las autoridades de salud pública, principalmente debido a sus características ambiguas (MORAES et al., 2012). Por un lado, contribuyen al desarrollo de propiedades sensoriales a través de reacciones bioquímicas durante la maduración, como la proteólisis, la lipólisis, el uso de citrato y la producción de compuestos aromáticos volátiles (HUGAS et al., 2003; JAMET et al., 2012). Por otro lado, se han convertido en patógenos importantes en pacientes hospitalizados y en personas mayores con enfermedades graves subyacentes (LECLERQ, 2009; ROSENTHAL et al., 2015; SIEVERT et al., 2013; ARCHAMBAUD et al., 2019). En los últimos años, se les ha asociado con bacteriemia, sepsis y endocarditis bacteriana (HEIDARI et al., 2017), y estas infecciones pueden originarse tanto en la microbiota normal como transmitirse de paciente a paciente o adquirirse a través del consumo de agua o alimentos contaminados (MURRAY et al., 2004).
Inicialmente, Enterococcus spp. se clasificó como Strept
ococcus del grupo D debido a que sus especies poseen el antígeno de la pared celular del grupo D asociado a su membrana citoplasmática (TEIXEIRA e MERQUIOR, 2013). Entre las más de 50 especies del género Enterococcus spp., E. faecalis y E. faecium se aíslan con mayor frecuencia en humanos, animales y muestras de alimentos. Del 80 al 90% de los Enterococcus spp. aislados de muestras clínicas en humanos son de la especie E. faecalis, el 5 al 15% son E. faecium, pero ocasionalmente se pueden identificar otras especies en estos aislamientos, como E. hirae, E. durans, E. gallinarum o E. casseliflavus (SILVA et al. Sin embargo, a diferencia de E. faecalis o E. faecium, E. durans no es un patógeno común en humanos (BYUNG et al., 2019).
RESISTENCIA ANTIMICROBIANA Y ENTEROCOCCUS SPP.
La aparición de resistencia antimicrobiana en enterococos y su propagación en alimentos sugieren un riesgo para la salud pública, y se debe considerar una posible correlación entre las cepas presentes en hospitales y las aisladas de alimentos (RIBOLDI et al., 2009). La aparición de la resistencia bacteriana multidroga en el entorno hospitalario ha aumentado en las últimas décadas, lo que ha llevado al aumento de la morbilidad y la mortalidad, el tiempo de hospitalización y, en consecuencia, los costos de atención médica. Esto representa un desafío para la salud pública y ha llevado a un aumento en la prescripción de antibióticos (LÍRIO et al., 2019). Entre los principales patógenos bacterianos hospitalarios resistentes a los antibióticos disponibles se encuentra el género Enterococcus, cuyas infecciones plantean verdaderos desafíos terapéuticos (BLANCO et al., 2016).
La resistencia antimicrobiana (RAM) está aumentando en todo el mundo y amenaza la prevención y el tratamiento de infecciones. En 2016, la resistencia antimicrobiana causó 70,000 muertes y se estima que para 2050 causará hasta 10 millones de muertes al año. Esta estimación se basa en la realidad actual de un uso excesivo de antibióticos, tratamientos incompletos, falta de control de infecciones, saneamiento deficiente en los países en desarrollo y la globalización, que facilita la distribución de microorganismos por todo el mundo. Estos factores contribuyen a la RAM, que, combinada con la escasa producción de nuevos antibióticos, da como resultado infecciones sin tratamiento (BELLO y DINGLE, 2018).
Se sabe que Enterococcus spp. tiende a experimentar selección natural con cada aplicación de antimicrobianos, lo que lleva a la formación de un reservorio animal de Enterococcus spp.
La resistencia genética a sustancias dañinas es un proceso de protección natural en algunas especies bacterianas que, en algunos casos, se ha predeterminado durante millones de años (BARLOW y HALL, 2002; LEBRETON et al., 2017). Sin embargo, el uso excesivo e indiscriminado de antibióticos, tanto en el tratamiento de infecciones humanas como en la ganadería, ha resultado en un aumento exponencial en las tasas de resistencia. En la actualidad, la resistencia bacteriana se considera uno de los mayores problemas de salud pública relacionados con la morbilidad y la mortalidad (OLIVEIRA, 2019).
Se sugiere que el uso de antimicrobianos en la alimentación animal como promotores del crecimiento ha creado grandes reservorios de genes de resistencia a antibióticos en varios ecosistemas y, por lo tanto, una posible vía de transmisión de Enterococcus spp. a través de la cadena alimentaria (SHEPARD y GILMORE, 2002).
El género Enterococcus spp. tiene relevancia clínica no solo debido a su creciente prevalencia en las últimas décadas, sino también debido a la alta incidencia de cepas resistentes a antibióticos (MEDEIROS, 2011). Riboldi et al. (2009), al analizar alimentos y productos lácteos “naturales” en Porto Alegre, RS, Brasil, observaron una alta frecuencia de resistencia a antimicrobianos utilizados en la agricultura, y también encontraron incidencia de resistencia a la nitrofurantoína, un antimicrobiano utilizado en el tratamiento de infecciones genitourinarias, en aislamientos de queso y repollo.
A pesar de revolucionar las prácticas médicas y ser uno de los mayores avances del último siglo, los agentes antimicrobianos tienen un período limitado de utilidad clínica debido a la aparición de resistencia (LUO et al., 2019).
El aumento de la resistencia antimicrobiana es un problema creciente, no solo en entornos hospitalarios, sino también para pacientes no hospitalizados (PÓVOA et al., 2019), ya que afecta significativamente la prevención y el tratamiento efectivo de una gran cantidad de infecciones causadas por bacterias, parásitos, virus y hongos (CAMACHO et al., 2018).
El uso inadecuado de antimicrobianos puede empeorar una infección y llevar al desarrollo de resistencia bacteriana en su tratamiento (OLIVEIRA y OLIVEIRA, 2014). Reflexionando más profundamente sobre el tema, se sabe que existe resistencia natural, lo cual es importante, pero la actitud humana de usar antimicrobianos de manera racional puede revertir este aspecto de la resistencia, beneficiando principalmente la salud pública (CAMACHO et al., 2018).
Uno de los problemas de salud pública más graves, la resistencia antimicrobiana, tiene una gran relevancia clínica, ya que dificulta el control de las enfermedades infecciosas cuando disminuye la eficacia del tratamiento, lo que lleva al aumento de las tasas de morbilidad y mortalidad. Además, genera costos significativos en el sistema de salud y puede promover la transmisión de infecciones a otros individuos (MONTEMAYOR et al., 2014). En los servicios de atención médica, especialmente en países en desarrollo, existe un grave problema de salud que es la aparición de resistencia bacteriana a los antibióticos (PEREIRA et al., 2013).
Los mecanismos de resistencia a los antibióticos de muchos microorganismos son diversos, con aspectos fisiopatológicos que involucran enzimas como las β-lactamasas, que degradan las penicilinas y algunas cefal
osporinas, otras enzimas modifican y desactivan el cloranfenicol y los aminoglucósidos, como la estreptomicina y la gentamicina. Otro mecanismo de resistencia se relaciona con la forma en que se transporta el antibiótico, como la resistencia a las tetraciclinas, el cloranfenicol y las fluoroquinolonas. Un tercer tipo de mecanismo altera el fármaco dentro de la célula, como el ribosoma, enzimas metabólicas o proteínas involucradas en la replicación del ácido desoxirribonucleico (ADN) o en la síntesis de la pared celular, lo que evita que el antibiótico inhiba una función vital en la célula microbiana. Pueden existir más de un tipo de mecanismo que proporcione resistencia al mismo antibiótico (NIKAIDO, 1996; LEVY y MARSHALL, 2004).
El crecimiento de Enterococcus spp. resistentes a la vancomicina (ERV) en entornos hospitalarios es una gran preocupación, ya que los ERV tienen la capacidad de transmitir su resistencia a otros microorganismos, incluido el Staphylococcus aureus resistente a la meticilina (MRSA), por lo que se monitorea e investiga de cerca en las unidades hospitalarias (FARON et al., 2016).
Según Terra et al. (2018), la resistencia antimicrobiana en Enterococcus spp. ocurre principalmente mediante cambios en el sitio de acción antimicrobiana y puede ocurrir mediante diferentes mecanismos: cambio en la pared celular, cambio en la membrana celular, cambio en la síntesis de proteínas, inhibición de la síntesis de ADN, inhibición competitiva de la síntesis de ácido fólico e inhibición de la síntesis de ácido ribonucleico (ARN).
Según el Centro para el Control y la Prevención de Enfermedades (CDC) (2019), durante muchos años, la única opción terapéutica para tratar las infecciones causadas por Staphylococcus aureus resistente a la meticilina era la vancomicina. El gen de resistencia a la vancomicina mediado por plásmidos, vanA, se encuentra regularmente en aislados resistentes a la vancomicina asociados con la atención médica para enterococos. Por lo tanto, existe una gran oportunidad para la transferencia de genes de resistencia a la vancomicina a S. aureus en pacientes o lugares donde ambos organismos coexisten. En los últimos 15 años, han surgido nuevas drogas como daptomicina, linezolid y oritavancina para el tratamiento de la resistencia a infecciones por Gram positivos. Aunque la resistencia a estas drogas se ha confirmado en algunos casos, tienen una actividad considerablemente alta contra Staphylococcus aureus resistente a la meticilina y tienen el potencial de tratar Staphylococcus aureus resistente a la vancomicina.
Los sistemas estandarizados para monitorear el uso de antibióticos son un requisito esencial como parte de una estrategia de control de la resistencia a los antibióticos. El uso de antibióticos puede cuantificarse en entornos hospitalarios para comprender los costos, la sensibilidad y la susceptibilidad rutinaria a los antibióticos en cada servicio de salud (TERÁN et al., 2018). Tres aspectos importantes para el éxito en la lucha contra la resistencia antimicrobiana, según la literatura, serían optimizar la administración de los agentes antimicrobianos existentes, prevenir la transmisión de organismos resistentes a medicamentos mediante el control de infecciones y reducir la contaminación ambiental (DAVEY et al., 2017).
INFECCIÓN NOSOCOMIAL Y ENTEROCOCCUS SPP.
Según la Ordenanza nº 2616/1998 del Ministerio de Salud (MS) (BRASIL, 1998), la infección hospitalaria es aquella adquirida después de la admisión del paciente y que se manifiesta durante la internación o después del alta, cuando puede estar relacionada con la internación o con procedimientos. Según Monteiro (1993), esta infección puede definirse como cualquier infección relacionada con la internación en establecimientos de salud, donde el paciente no presentó la patología durante el período de incubación en el momento de la admisión al hospital, a menos que haya habido una internación previa relacionada con ella.
Las Infecciones Hospitalarias (IH) son un problema de salud pública debido a la morbimortalidad que pueden causar, así como a los costos sociales y económicos asociados a ellas. Pueden definirse como enfermedades que afectan a los pacientes durante la internación o después del alta. El Centro para el Control y la Prevención de Enfermedades (CDC) enfatiza que la infección no debe estar presente durante el período de incubación durante la internación ni relacionada con internaciones anteriores (COSTA et al., 2019).
La presencia de determinantes genéticos que codifican mecanismos de resistencia a los antimicrobianos utilizados en la clínica médica es la base para la capacidad de colonizar y persistir en ambientes hospitalarios (TERRA et al., 2017). A pesar de formar parte del tracto gastrointestinal, Enterococcus spp. se consideran agentes oportunistas que con frecuencia causan infecciones hospitalarias.
Entre las especies que más causan patogénesis se encuentran Enterococcus faecalis con una tasa del 90% y Enterococcus faecium con un 10% (KRAHN y FACHINETTO, 2019). Los enterococos se consideran patógenos oportunistas que a menudo causan infecciones en pacientes internados durante largos períodos y/o tratados con varios antibióticos (REIS et al., 2001), y estas infecciones pueden ocurrir en el tracto gastrointestinal, el tracto urinario, las lesiones cutáneas, el corazón y la sangre (KAO y KLINE, 2019).
Enterococcus spp. como especies tienen una amplia capacidad de adaptación, lo que facilita su colonización en ambientes hospitalarios y puede llevar a la contaminación del paciente (LEBRETON et al., 2014). Dado que es un microorganismo oportunista, es preocupante que en los últimos años los estudios hayan mostrado un aumento gradual en la detección de factores de virulencia en cepas de Enterococcus spp. aisladas de fuentes clínicas, lo que también ha llevado a la observación de estos genes de virulencia en cepas aisladas de muestras de alimentos (OMS, 2017).
Según Teixeira (2019), la formación de biofilm en dispositivos hospitalarios, como resultado de la capacidad de adhesión de los microorganismos a las superficies, ha llevado a un crecimiento continuo de infecciones nosocomiales que representan un problema de salud pública. Además, según Fleming et al. (2016) y Omar et al. (2017), la resistencia antimicrobiana puede aumentar posiblemente en los biofilms como resultado de la propagación de genes de resistencia entre las células mediante la transferencia horizontal de genes facilitada por la proximidad de las células del biofilm.
BIOFILME – FORMACIÓN
Un biofilme es una estructura compuesta por un conjunto de microorganismos envueltos en una matriz extracelular de exopolisacáridos poliméricos que se adhieren a una superficie sólida (SHIRTLIFF et al., 2002; COSTERTON et al., 1999). El término “biofilme” surgió para describir la forma de vida microbiana sésil, definida por la adhesión de microorganismos a superficies sólidas, con la consiguiente producción de sustancias poliméricas extracelulares que forman una red gelatinosa que inmoviliza y protege a las células. La formación de biofilmes provoca cambios fenotípicos en las células planctónicas, que pueden describirse como estrategias de supervivencia de los microorganismos en entornos con condiciones adversas (COSTERTON et al., 1999).
Los microorganismos en entornos naturales que existen en forma de biofilmes pueden detectarse en la gran mayoría de los sustratos que tienen la humedad suficiente para mantener su crecimiento, con una tasa de detección entre el 95% y el 99%. En cuanto a la composición del biofilme, se observa que el agua es la fracción más significativa, llegando a representar hasta el 97% de la matriz del biofilme (OLIVEIRA et al., 2010). Se investigan intensamente no solo por sus efectos en la salud y el medio ambiente, sino también porque tienen un enorme potencial como herramientas para procesos biotecnológicos (HANSEN et al., 2019).
La capacidad de formar biofilme es una característica importante de virulencia de las bacterias (DONELLI y GUAGLIANONE, 2004). Por lo tanto, la detección de cepas productoras de biofilme es de gran importancia para establecer políticas de control, ya que las fallas en el proceso de limpieza pueden permitir que los residuos adheridos a equipos y superficies se conviertan en una fuente potencial de contaminación en la industria de alimentos y en entornos hospitalarios (CASSENEGO et al., 2013).
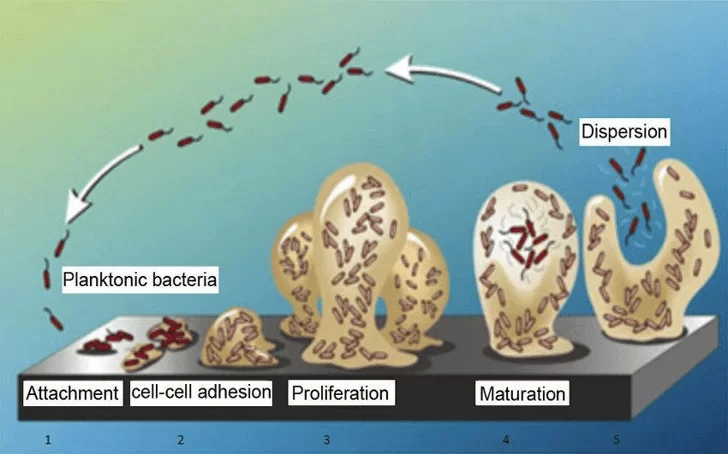
La formación del biofilme bacteriano se puede dividir en cuatro etapas. Primero, hay una adhesión inicial de las células bacterianas, seguida de la agregación y formación de múltiples capas de células, luego la maduración del biofilme y, finalmente, la liberación de las células del biofilme para iniciar un nuevo ciclo de formación de biofilme en otro lugar (Figura 01). Las interacciones iniciales que ocurren entre las bacterias y la superficie son inespecíficas y se impulsan mediante diferentes fuerzas: fuerzas hidrofóbicas, electrostáticas, fuerzas de Lifshitz-Van Der Waals, entre otras (GUTIÉRREZ et al., 2012; SÁNCHEZ et al., 2014).
Con el inicio de la maduración celular, las células comienzan a dividirse y a formar pequeños agregados celulares caracterizados por la ocurrencia del Quorum Sensing (QS) (FONSECA, 2011), que es un mecanismo de comunicación entre las bacterias a través de la producción y difusión de pequeñas moléculas químicas o señalizadoras. El QS permite la coordinación del comportamiento bacteriano en relación con el entorno mediante la regulación de la expresión de genes especializados en respuesta a la densidad poblacional (LAZAR, 2011; PINTO y FONSECA, 2018).
Figura 01. Las cuatro etapas de la formación del biofilme bacteriano en superficies
Según Costa et al. (2018), la industria de procesamiento de alimentos lácteos tiene un gran problema con la formación de biofilm, que generalmente está asociada a serios problemas de higiene que pueden causar deterioro y recontaminación del producto. Por otro lado, según Bosman et al. (2014), una de las principales preocupaciones de la medicina moderna es la infección asociada al biofilm dentro del entorno hospitalario, ya que su incidencia ha aumentado debido al creciente uso de dispositivos médicos implantables, lo que ha contribuido directamente a tasas elevadas de morbilidad y mortalidad en pacientes hospitalizados.
FORMACIÓN DE BIOFILM POR ENTEROCOCCUS SPP.
Enterococcus spp. asociados a brotes de origen alimentario poseen factores de virulencia, como la capacidad de formar biofilm. La agregación microbiana por esta especie bacteriana aparentemente está influenciada por varios aspectos, como la temperatura, la exposición a nutrientes y desinfectantes, y las características de la superficie (COSTA et al., 2018).
Los enterococos aislados de alimentos que muestran la capacidad de formar biofilm son preocupantes, ya que esta capacidad contribuye a la supervivencia, persistencia y propagación de enterococos resistentes y/o genes de resistencia en diversas condiciones ambientales (MEDEIROS et al., 2014).
Según Estrela et al. (2009), las fases de organización estructural del biofilm, la composición y las actividades de los microorganismos colonizadores en varios entornos pueden diferir, aunque el establecimiento de una microcomunidad en una superficie parece seguir esencialmente la misma serie de etapas de desarrollo.
Según Medeiros (2011), se ha demostrado que varios factores pueden influir en la formación de biofilm en Enterococcus spp., además de las condiciones que generalmente afectan la formación de biofilm en varios microorganismos, como el pH, la concentración de CO2, la temperatura, entre otros. Aún no hay consenso sobre la influencia de la concentración de glucosa en la formación de biofilm por Enterococcus spp., pero algunos investigadores creen que la glucosa aumenta la producción de biofilm por Enterococcus spp.
Entre las especies de este género, Enterococcus faecalis es un patógeno oportunista que causa la mayoría de las infecciones por enterococos con la capacidad de adherirse a superficies bióticas y abióticas, promoviendo la formación de un biofilm que le permite crecer y sobrevivir en entornos hostiles (CÂNDIDO et al., 2010).
Entre los diversos factores involucrados en la patogénesis de estos organismos, la formación de biofilm y la resistencia a los antibióticos son los más importantes. La capacidad de formar biofilm ha sido atribuida a la presencia de algunos genes de virulencia (SAFFARI et al., 2017); por lo tanto, se observan otros mecanismos de control de la infección, como la prevención de la formación de biofilm por Enterococcus spp. o la inhibición de la acción de otros factores de virulencia, lo que podría proporcionar un método alternativo de terapia, especialmente teniendo en cuenta que el tratamiento antimicrobiano es un desafío para este género (COMERLATO et al., 2013). La formación robusta de biofilm y la resistencia inherente a múltiples drogas en los enterococos los convierten en patógenos nosocomiales desafiantes (SURYALETHA et al., 2019).
CONCLUSIÓN
Los Enterococcus son microorganismos versátiles que pueden encontrarse como contaminantes en el queso de coalho y causar intoxicaciones alimentarias. Han surgido como una de las bacterias Gram-positivas más prevalentes en infecciones nosocomiales, principalmente debido a su resistencia a múltiples antimicrobianos. Los estudios sugieren graves problemas de salud pública, ya que estos microorganismos pueden tener la capacidad de transferir genes de resistencia antimicrobiana a otros microorganismos presentes en la microbiota intestinal humana y animal, lo que hace imposible el uso de medicamentos para el tratamiento clínico estándar mencionado en el artículo. Se necesitan más estudios y análisis de Enterococcus en alimentos y su capacidad para resistir a agentes antimicrobianos para comprender los mecanismos de transferencia de resistencia entre estos microorganismos.
REFERENCIAS
ALI, S. A. et al. Environmental Enterococci: I. Prevalence of virulence, antibiotic resistance and species distribution in poultry and its related environment in Karachi, Pakistan. Letters in Applied Microbiology. v. 58, n. 5, p. 423-432, 2014. Disponível em: <10.1111/lam.12208>. Acesso em: 2 jun. 2023.
ANDRADE, A. A. Estudo do perfil sensorial, físico-químico e aceitação de queijo de coalho produzido no estado do Ceará. 2006. Dissertação (Mestrado em Tecnologia de Alimentos) – Centro de Ciências Agrárias, Universidade Federal do Ceará, Fortaleza. Disponível em: <http://www.repositorio.ufc.br/handle/riufc/17217>. Acesso em: 2 jun. 2023.
ANDRADE, S. D. E. Caracterização fenotípica e genotípica de amostras clínicas e indígenas de Enterococcus isoladas de seres humanos: diversidade, virulência e resistência a drogas antimicrobiana. 2018. Tese (Doutorado em Ciências Biológicas (Microbiologia)) – Universidade Federal de Minas Gerais, Belo Horizonte – MG. Disponível em: <http://hdl.handle.net/1843/BUOS-B76JMH>. Acesso em: 2 jun. 2023.
ARCHAMBAUD, C. et al. Intestinal translocation of enterococci requires a threshold level of enterococcal overgrowth in the lumen. Scientific Reports. v. 9, n. 8926, pp. 1-12, 2019. Disponível em: <https://doi.org/10.1038/s41598-019-45441-3>. Acesso em: 2 jun. 2023.
ARIAS, C. A.; MURRAY, B. E. The rise of the Enterococcus: Beyond vancomycin resistance. Nature Reviews Microbiology. v. 10, n. 4, pp. 266-278, 2012. Disponível em: <10.1038/nrmicro2761>. Acesso em: 6 jun. 2023.
BARLOW, M.; HALL, B. G. Phylogenetic analysis shows that the OXA beta-lactamase genes have been on plasmids for millions of years. Journal of Molecular Evolution. v. 55, n. 3, pp. 314-321, 2002. Disponível em: <10.1007/s00239-002-2328-y>. Acesso em: 6 jun. 2023.
BELLO, A.; DINGLE, T. C. What’s that resistance mechanism? Understanding genetic determinants of gram-negative bacterial resistance. Clinical Microbiology Newsletter. v. 40, n. 20, pp 165-174, 2018. Disponível em: <10.1016/j.clinmicnews.2018.10.001>. Acesso em: 6 jun. 2023.
BLANCO, A. L.; FREITAS, L. M.; FONTANA, C. R. ACT. Avaliação de resistência à Terapia Fotodinâmica em Enterococcus faecalis. Revista de Ciências Farmacêuticas Básica e Aplicada. v. 37, n. 1, pp. 1-165, 2016. Disponível em: <file:///C:/Users/Workstation%20Video%20B/Downloads/618-Article%20Text-1879-2-10-20200221.pdf>. Acesso em: 6 jun. 2023.
BYUNG-HAN, R. et al. Clinical characteristics and treatment outcomes of Enterococcus durans bacteremia: a 20-year experience in a tertiary care hospital. European Journal of Clinical Microbiology & Infectious Diseases. v. 38, n. 9, pp. 1743-1751, 2019. Disponível em: <https://pubmed.ncbi.nlm.nih.gov/31243595/>. Acesso em: 6 jun. 2023.
BORGES, M. F. et al. Microrganismos patogênicos e indicadores em queijo de coalho produzido no Estado do Ceará, Brasil. Boletim do Centro de Pesquisa de Processamento de Alimentos. v. 21, n. 1, pp. 31- 40, 2003. Disponível em: <10.5380/cep.v21i1.1146>. Acesso em: 6 jun. 2023.
BOSMAN, W. M. P. F. et al. Infections of intravascular bare metal stents: a case report and review of literature. European Journal of Vascular and Endovascular Surgery. v. 47, n. 1, pp. 87-99, 2014. Disponível em: < https://pubmed.ncbi.nlm.nih.gov/24239103/>. Acesso em: 6 jun. 2023.
BRASIL. Ministério da Saúde. Agência Nacional de Vigilância Sanitária – ANVISA. Portaria n. 2616 de 12 de maio de 1998. Brasília. Disponível em <https://bvsms.saude.gov.br/bvs/saudelegis/gm/1998/prt2616_12_05_1998.html>. Acesso em 19/07/2019.
BRASIL. Ministério da Agricultura. Secretaria de Defesa Agropecuária. Departamento de Inspeção de Produtos de Origem Animal DIPOA. Portaria n. 146 de 7 de março de 1996. Aprova os Regulamentos Técnicos de Identidade e Qualidade de Produtos Lácteos. Diário Oficial da União, Brasília, 11 de março de 1996.
CAMACHO, J. O. I. et al. Prescripción racional de antibióticos: una conducta urgente. Medicina Interna de México. v. 34, n. 5, pp. 762-770, 2018. Disponível em: <https://pesquisa.bvsalud.org/portal/resource/pt/biblio-984739>. Acesso em: 7 Jun. 2023.
CÂNDIDO, C. S. et al. Effects of Myrcia ovata Cambess. Essential oil on planktonic growth of gastrointestinal microorganisms and biofilm formation of Enterococcus faecalis. Brazilian Journal of Microbiology. v. 41, n. 3, pp. 621-627, 2010. Disponível em: <https://pesquisa.bvsalud.org/portal/resource/pt/lil-549403>. Acesso em: 7 Jun. 2023.
CASSENEGO, A. P. V.; ELLWANGER, J.; PEDRO, A.; AZEVEDO, A.; RIBEIRO, M.L.; FRAZZON, A. P. G. Virulência e formação de biofilme microbiano por Enterococcus faecalis isolados de swabs cloacais de frangos de corte infectados com Eimeria spp. Pesquisa Veterinária Brasileira. v. 33, n. 12, pp. 1433-1440 2013. Disponível em: <https://www.scielo.br/j/pvb/a/PsDXt5fJyrS6gjZ9xCtFqfw/?lang=pt>. Acesso em: 7 Jun. 2023.
CENTERS FOR DISEASE CONTROL AND PREVENTION, C.D.C. (online). Antibiotic resistance threats in the United States. 2019. Disponível em:
<https://www.cdc.gov/drugresistance/pdf/threats-report/2019-ar-threats-report-508.pdf>. Acesso em: 22 jul. 2019.
COMERLATO, C. B. et al. Presence of virulence factors in Enterococcus faecalis and Enterococcus faecium susceptible and resistant to vancomycin. Memória do Instituto Oswaldo Cruz. v. 108, n. 5, pp. 590-595, 2013. Disponível em: <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3970601/>. Acesso em: 7 Jun. 2023.
COSTA, F. T. Síntese, caracterização e o estudo do efeito de nano-partículas de CoFe2O4-YFe2O3 em bactérias patogênicas. 2013. Dissertação (Mestrado em Ciências de Materiais) – Universidade de Brasília, Brasília. Disponível em: <https://repositorio.unb.br/handle/10482/14397>. Acesso em: 7 Jun. 2023.
COSTA, G. A. et al. Evaluation antibacterial and antibiofilm activity of the antimicrobial peptide P34 against Staphylococcus aureus and Enterococcus faecalis. Anais Academia Brasileira de Ciências. v. 90, n. 1, pp. 1-12, 2018. Disponível em: <https://doi.org/10.1590/0001-3765201820160131>. Acesso em: 7 Jun. 2023.
COSTA, M. C. P. et al. Micro-Organismos isolados a partir de espécimes clínicos de centro cirúrgico. Revista Saúde e Desenvolvimento. v. 13, n. 14, pp. 1-21, 2019. Disponível em: <https://www.revistasuninter.com/revistasaude/index.php/saudeDesenvolvimento/article/view/1013>. Acesso em: 7 Jun. 2023.
COSTERTON, J. W.; STEWART, P. S.; GREENBERG, E. P. Bacterial biofilms: a common cause of persistent infections. Science. v. 284, n. 5418, p. 1318-1322, 1999. Disponível em: <https://www.science.org/doi/10.1126/science.284.5418.1318>. Acesso em: 7 Jun. 2023.
DAVEY, P. et al. Interventions to improve antibiotic prescribing practices for hospital inpatients. Cochrane Database of Systematic Reviews. v. 2, n. 2, pp. 1-326, 2017. Disponível em: <https://doi.org/10.1002/14651858.CD003543.pub4>. Acesso em: 7 Jun. 2023.
DERDE, L. P.; DAUTZENBERG, M. J.; BONTEN, M. J. Chlorhexidine body wasshing to control antimicrobial-resistant bacteria in intensive care units: a systematic review. Intensive Care Medicine. v. 38, n. 6, pp. 931-939, 2012. Disponível em: <10.1007/s00134-012-2542-z>. Acesso em: 7 Jun. 2023.
DONELLI, G.; GUAGLIANONE, E. Emerging role of Enterococcus spp. In catheter related infections: biofilm formation and novel mechanisms of antibiotic resistance. The Journal of Vascular. v. 5, n. 1, pp. 3-9, 2004. Disponível em: <10.1177/112972980400500101>. Acesso em: 7 Jun. 2023.
ESTRELA, C. R. A. et al. model system to study antimicrobial strategies in endodontic biofilms. Journal of Applied Oral Science. v. 17, n. 2, 2009. Disponível em: <https://www.scielo.br/j/jaos/a/rzGrQDfDvY7RJMJkMwfJPTx/?lang=en>. Acesso em: 7 Jun. 2023.
FARON, M. L.; LEDEBOER, N. A.; BUCHAN, B. W. Resistance mechanisms, epidemiology, and approaches to screening for vancomycin-resistant Enterococcus in the health care setting. Journal of Clinical Microbiology. v. 54, n. 10, pp. 2436- 2447, 2016. Disponível em: <10.1128/JCM.00211-16>. Acesso em: 7 Jun. 2023.
FISHER, K.; PHILLIPS, C. The ecology, epidemiology and virulence of Enterococcus. Microbiology. v. 155, n. 6, pp. 1749-1757, 2009. Disponível em: <10.1099/mic.0.026385-0>. Acesso em: 7 Jun. 2023.
FLEMMING, H. C. et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. v. 14, n. 9, pp. 563-575, 2016. Disponível em: <https://www.nature.com/articles/nrmicro.2016.94>. Acesso em: 7 Jun. 2023.
FONSECA, A. P. Biofilms in wounds: An unsolved problem? Working together to ensure better patient outcomes. EWMA Journal. V. 11, n. 2, p. 10- 23, 2011.
FREITAS FILHO, J. R. et al. Avaliação da qualidade do queijo “coalho” artesanal fabricado em Jucati-PE. Revista Eletrônica de Extensão – Extensio. v. 6, n. 8, p. 35-49, 2009. Disponível em: <https://periodicos.ufsc.br/index.php/extensio/article/view/1807-0221.2009v6n8p35/11446>. Acesso em: 7 Jun. 2023.
GIRAFFA, G. Functionality of enterococci in dairy products. International Journal of Food Microbiology. v. 88, n. 2-3, p. 215-222, 2003. Disponível em: <10.1016/s0168-1605(03)00183-1>. Acesso em: 7 Jun. 2023.
GIRAFFA, G; ROSSETTI, L. Monitoring of the bacterial composition of dairy starter cultures by RAPD-PCR. FEMS Microbiology Letters. v. 237, n. 1, p. 133-138, 2004. Disponível em: <https://doi.org/10.1111/j.1574-6968.2004.tb09688.x>. Acesso em: 7 Jun. 2023.
GUTIÉRREZ, D. et al. Incidence of Sthapylococcus aureus and analysis of associated bacterial communities on food industry surfaces. Applied and Environmental Microbiology. v. 78, n. 24, p. 8547-8554, 2012. Disponível em: <10.1128/AEM.02045-12>. Acesso em: 7 Jun. 2023.
HANSEN, S. H. et al. Machine-assisted cultivation and analysis of biofilms. Scientific Reports. v. 9, n. 8933, pp. 1-10, 2019. Disponível em: <file:///C:/Users/Workstation%20Video%20B/Downloads/s41598-019-45414-6.pdf>. Acesso em: 7 Jun. 2023.
HEIDARI, H. et al. High Incidence of Virulence Factors among Clinical Enterococcus faecalis Isolates In Southwestern Iran. Infect Chemother. v. 49, n. 1, p. 51-56, 2017. Disponível em: <10.3947/ic.2017.49.1.51>. Acesso em: 7 Jun. 2023.
HOLLENBECK, B. L.; RICE, L. B. Intrinsic and acquired resistance mechanisms in Enterococcus. Virulence. v. 3, n. 5, p. 421-433, 2012. Disponível em: <10.4161/viru.21282>. Acesso em: 7 Jun. 2023.
HUGAS, M.; GARRIGA, M. M.; AYMERICH, M. T.; Functionality of enterococci in meat products. International Journal of Food Microbiology. v. 88, n. 2, p. 223-233, 2003. Disponível em: <10.1016/s0168-1605(03)00184-3>. Acesso em: 9 Jun. 2023.
IBGE – INSTITUTO BRASILEIRO DE GEOGRAFIA E ESTATÍSTICA. Censo Agropecuário 2017. Rio de Janeiro: IBGE, 2017. Disponível em: <https://sidra.ibge.gov.br/pesquisa/censo-agropecuario/censo-agropecuario-2017/resultados-definitivos>. Acesso em: 22 jun. 2019.
IBGE – INSTITUTO BRASILEIRO DE GEOGRAFIA E ESTATÍSTICA. Produção da Pecuária Municipal 2018. Rio de Janeiro: IBGE, v. 46, p. 1-8, 2018. Disponível em: <https://biblioteca.ibge.gov.br/visualizacao/periodicos/84/ppm_2018_v46_br_informativo.pdf>. Acesso em: 9 fev. 2020.
JAMET, E. et al. Prevalence and characterization of antibiotic resistant Enterococcus faecalis in French cheeses. Food Microbiology. v. 31, n. 2, p.191-198, 2012. Disponível em: <10.1016/j.fm.2012.03.009>. Acesso em: 7 Jun. 2023.
KAO, P. H. N.; KLINE, K. A. Dr. Jekyll and Mr. Hide: How Enterococcus faecalis Subverts the Host Immune Response to Cause Infection. Journal of Molecular Biology. v. 431, n. 16, p. 2932- 2945, 2019. Disponível em: <10.1016/j.jmb.2019.05.030>. Acesso em: 9 Jun. 2023.
KARAGULER, T. KAHRAMAN, H. TUTER, M. Analyzing effects of ELF electromagnetic fields on removing bacterial biofilm. Biocybernetics and Biomedical Engineering. v. 37, n. 2, p. 336-340, 2017. Disponível em: <10.1016/j.bbe.2016.11.005>. Acesso em: 01 maio 2023.
KASZANYITZKY, E. J.; TENK, M.; GHIDÁN, A.; FEHÉVÁRI, G. Y.; PAPP, M. Antimicrobial susceptibility of enterococci strains isolated from slaughter animals on the data of Hungarian resistance monitoring system from 2001 to 2004. International Journal of food microbiology. v.115, n. 1, p. 119-123, 2007. Disponível em: <10.1016/j.ijfoodmicro.2006.10.004>. Acesso em: 7 Jun. 2023.
KONEMAN, E. W. et al. Enterococcus. In: KONEMAN, Elmer W. (Ed.). Diagnóstico microbiológico. 7. ed. Rio de Janeiro: Guanabara Koogan. p. 589-659, 2014.
KRAHN, C. O.; FACHINETTO, J. M. Enterococcus spp.: Características gerais e considerações médicas, uma revisão. 6º Congresso Internacional em Saúde. 2019. Disponível em: <file:///C:/Users/Workstation%20Video%20B/Downloads/10765-Texto%20do%20artigo-41799-1-10-20190424.pdf>. Acesso em: 7 Jun. 2023.
LÁCTEA BRASIL. Queijo – Alimento nobre e saudável. [S. l.: s. n.], 2006. Disponível em: <https://www.yumpu.com/pt/document/read/13019755/queijo-alimento-nobre-e-saudavel-nobre-e-saudavel-capril-virtual>. Acesso em 30 jul. 2019.
LAZAR, V. Quorum sensing in biofilms – how to destroy the bacterial citadels or their cohesion/power? Anaerobe. v.17, n. 6, p. 280-285, 2011. Disponível em: <10.1016/j.anaerobe.2011.03.023>. Acesso em: 7 Jun. 2023.
LEBRETON, F. et al. Tracing the Enterococci from Paleozoic Origins to the Hospital. Cell. v. 169, n. 5, p. 849-861, 2017. Disponível em: <10.1016/j.cell.2017.04.027>. Acesso em: 7 Jun. 2023.
LEBRETON, F. et al. Enterococcus diversity, origins in nature, and gut colonization/ Enterococci: from commensals to leading causes of drug resistant infection. Boston: Massachusetts Eye and Ear Infirmary. p.1-59, 2014. Disponível em: <https://www.ncbi.nlm.nih.gov/books/NBK190427/>. Acesso em: 7 Jun. 2023.
LECLERCQ, R. Epidemiological and resistance issues in multidrug-resistant staphylococci and enterococci. Clinical Microbiology and Infection. v. 15, n. 3, p. 224-231, 2009. Disponível em: <10.1111/j.1469-0691.2009.02739.x>. Acesso em: 7 Jun. 2023.
LEVY, S.; MARSHALL, B. Antimicrobial resistance worlwide: causes, challenges and responses. Nature medicine supplement. v. 10, n. 12, p. 122-129, 2004. Disponível em: <https://www.nature.com/articles/nm1145>. Acesso em: 9 Jun. 2023.
LÍRIO, M.; ANDRADE, T.; MENDES, A. V.; BARBERINO, M. G. Avaliação da colonização por bactérias multirresistentes em pacientes admitidos via central de regulação do estado em um hospital filantrópico em Salvador, Bahia. Revista de Epidemiologia e Controle de Infecção. v. 9, n. 1, 2019. Disponível em: <https://online.unisc.br/seer/index.php/epidemiologia/article/view/11595>. Acesso em: 9 Jun. 2023.
LOSSOUARN, J. et al. Enterococcus faecalis Countermeasures Defeat a Virulent Picovirinae Bacteriophage. Journal viruses. v. 11, n. 1, p. 48, 2019. Disponível em: <https://doi.org/10.3390/v11010048>. Acesso em: 9 Jun. 2023.
LUNA, R. O. Identificação, perfil de resistência a antimicrobianos e caracterização molecular de Aeromonas spp. isoladas de queijos coalho tipo A, comercializados na cidade de Recife PE. 2012. Dissertação (Mestrado em Ciência Veterinária). UFRPE, Pernambuco – Recife.
LUO, X. et al. A diversity-oriented rhodamine library for wide-spectrum bactericidal agents with low inducible against resistant pathogens. Nature Communications. v. 10, n. 258, 2019. Disponível em: <https://www.nature.com/articles/s41467-018-08241-3>. Acesso em: 9 Jun. 2023.
MEDEIROS, A. W. Avaliação dos fatores de virulência e a capacidade de formação de biofilme in vitro em isolados alimentares e clínicos de Enterococcus spp. e utilização de PCR-RFLP para a identificação de Enterococcus casseliflavus e Enterococcus gallinarum. 2011 Dissertação (Mestrado). Universidade Feevale, Rio Grande do Sul.
MEDEIROS, A. W.; PEREIRA, R. I.; OLIVEIRA, D. V.; MARTINA, P. D.; AZEVEDO, P. A.; VAN DER SAND, S.; FRAZZON, J.; FRAZZON, A. P. G. Molecular detection of virulence factors among food and clinical Enterococcus faecalis strains in South Brazil; Brazilian Journal of Microbiology. vol. 45, n.1, 2014. Disponível em: <http://dx.doi.org/10.1590/S1517- 83822014005000031>. Acesso em 22 Jul. 2019.
MENEZES, S. S. M. “Queijo de coalho-rei do balcão”: Expansão da produção alicerçada pela demanda dos migrantes sertanejos. Habitus. v. 11, n. 2, p. 143-158, 2013. Disponível em: <https://doi.org/10.18224/hab.v11.2.2013.143-158>. Acesso em: 7 Jun. 2023.
MONTEIRO, J. A. INFECÇÕES NOSOCOMIAIS. Alguns Aspectos. ACTA Médica Portuguesa. v. 6, p. 135-140, 1993. Disponível em: <https://silo.tips/download/infecoes-nosocomiais-alguns-aspectos>. Acesso em: 7 Jun. 2023.
MONTEMAYOR, J. C. G.; BOFARULL, A. M.; MOCHALES, F. B. Impacto de los movimientos migratorios en la resistencia bacteriana a los antibióticos. Revista Española de Salud Pública. v. 88, n. 6, 2014. Disponível em: <https://scielo.isciii.es/scielo.php?script=sci_arttext&pid=S1135-57272014000600014>. Acesso em: 7 Jun. 2023.
MONTOYA, R. R. et al. Variables associadas a costos em cuidados intensivos. Revista de la Asociación Mexicana de Medicina Crítica y Terapia Intensiva. v. 29, n. 3, p. 138-144, 2015. Disponível em: <https://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S0187-84332015000300003>. Acesso em: 7 Jun. 2023.
MORAES, P. M. et al. Bacteriocinogenic and virulence potential of Enterococcus isolates obtained from raw milk and cheese. Journal of Applied Microbiology. v. 113, p. 318-328, 2012. Disponível em: <https://www.locus.ufv.br/bitstream/123456789/13991/1/Moraes_et_al-2012-Journal_of_Applied_Microbiology.pdf>. Acesso em: 9 Jun. 2023.
MURRAY, P. et al. Microbiologia Médica. 4 ed. Rio de Janeiro: Guanabara Koogan, p. 220-223, 2004. Disponível em: <https://eu-ireland-custom-media-prod.s3-eu-west-1.amazonaws.com/Brasil/Downloads/03-12/ESAMPLE-9788535290363.pdf>. Acesso em: 9 Jun. 2023.
NIKAIDO, H. Multidrug efflux pumps of gram-negative bacteria. Journal of bacteriology. v. 178, n. 20, p. 5853-5859, 1996. Disponível em: <10.1128/jb.178.20.5853-5859.1996>. Acesso em: 7 Jun. 2023.
NOGUEIRA, J. G. A embalagem como fator de agregação de valor ao produto: Um estudo do segmento de queijos em Juiz de Fora. 2006. Dissertação (Mestrado Profissional em Sistemas de Gestão) – Centro Tecnológico, Universidade Federal Fluminense, Niterói.
OLIVEIRA, A. F.; OLIVEIRA FILHO, H. Perfil microbiológico e de resistência antimicrobiana no pé diabético infectado. Jornal Vascular Brasileiro. v. 13, n. 4, p. 289-293, 2014. Disponível em: <https://www.scielo.br/j/rcbc/a/rPHTb6ZvfYkgd9rcczXdYjf/?lang=pt>. Acesso em: 7 Jun. 2023.
OLIVEIRA, E. S. Emergência de Enterococcus spp. resistentes à vancomicina na cidade do Natal – RN. 2019. Dissertação (Mestrado em Ciências Biológicas), Universidade Federal do Rio Grande do Norte. Natal – RN.
OLIVEIRA, M. M. M.; BRUGNETA, D. F.; PICCOLI, R. H. Biofilmes microbianos na indústria de alimentos: uma revisão. Revista do Instituto Adolfo Lutz. v. 69, n. 3, p. 277-284, 2010. Disponível em: <http://www.ial.sp.gov.br/resources/insituto-adolfo-lutz/publicacoes/rial/10/rial69_3_completa/1289.pdf>. Acesso em: 7 Jun. 2023.
OLIVEIRA, M. et al. Antimicrobial resistance and in vitro biofilm-forming ability of enterococci from intensive and extensive farming broilers. Poultry Science. v. 89, n. 5, p. 1065- 1069, 2010. Disponível em: <10.3382/ps.2008-00436>. Acesso em: 7 Jun. 2023.
OMAR, A. et al. Microbial Biofilms and Chronic Wounds. Journal Microorganisms. v. 5, n. 1, pp. 1-15, 2017. Disponível em: <10.3390/microorganisms5010009>. Acesso em: 7 Jun. 2023.
PARTE, A. C. LPSN-list of prokaryotic names with standing in nomenclature. Nucleic Acids Research. v. 42, n. 1, p. 613-616, 2014. Disponível em: <10.1093/nar/gkt1111>. Acesso em: 7 Jun. 2023.
PEREIRA, F. G. F. et al. Characterization of infections related to health care in a neonatal intensive care unit. Enfermagem UERJ. v. 21, n. 1, 2013. Disponível em: <https://www.e-publicacoes.uerj.br/index.php/enfermagemuerj/article/view/6370/5899>. Acesso em: 7 Jun. 2023.
PINTO, G.; FONSECA, A. F. Biofilmes e Feridas Crônicas: Um potencial algoritmo. Journal of tissue regeneration & healing. 2018. Disponível em: <https://bdigital.ufp.pt/bitstream/10284/5816/1/PPG_25983.pdf>. Acesso em: 9 Jun. 2023.
PORTO, B. C et al. Determinantes de virulência em Enterococcus endógenos de queijo artesanal. Revista Ciência Agronômica. v. 47, n. 1, p. 69-76, 2016. Disponível em: <http://ccarevista.ufc.br/seer/index.php/ccarevista/article/view/4297>. Acesso em: 9 Jun. 2023.
PÓVOA, A. C. et al. J. R. Evolução da resistência bacteriana em infecção comunitária do trato urinário em idosos. Revista de Epidemiologia e Controle de Infecção. v. 9, n. 1, p. 08-14, 2019. Disponível em: <https://online.unisc.br/seer/index.php/epidemiologia/article/view/10468>. Acesso em: 9 Jun. 2023.
REIS, A. O.; CORDEIRO, J. C. R.; MACHADO, A. M. O.; SADER, H. S. In vitro antimicrobial activity of linezolid tested against vancomycin-resistant enterococci isolated in Brazilian hospitals. Brazilian Journal of Infectious Diseases. v. 5, n. 5, 2001. Disponível em: <https://www.scielo.br/j/bjid/a/rNpQ6tBdzyFHjVLktGDt6SK/?lang=en>. Acesso em: 9 Jun. 2023.
RIBOLDI, G. P.; FRAZZON, J.; AZEVEDO, P. A.; Antimicrobial resistance profile of Enterococcus spp. isolated from food in Southern Brazil. Brazilian Journal of Microbiology. v. 40, p. 125-128, 2009. Disponível em: <https://www.scielo.br/j/bjm/a/fzR4WH7LGmKh3QRwk8fW3HH/?lang=en>. Acesso em: 9 Jun. 2023.
ROSENTHAL, V. D. et al. International nosocomial infection control consortium (INICC) report, data summary of 43 countries for 2007-2012. Device-associated module. American Journal of Infection Control. v. 43, n. 7, pp. 779-781, 2015. Disponível em: <https://pubmed.ncbi.nlm.nih.gov/25179325/>. Acesso em: 9 Jun. 2023.
SAFFARI, F.; DALFARDI, M. F.; MANSOURI, S.; AHMADRAJABI, R. Survey for Correlation between Biofilm Formation and Virulence Determinants in a Collection of Pathogenic and Fecal Enterococcus faecalis Isolates. Infection & chemotherapy. v. 49, n. 3, p. 176-183, 2017. Disponível em: <10.3947/ic.2017.49.3.176>. Acesso em: 9 Jun. 2023.
SÁNCHEZ, D. V.; CABO, M. L.; IBUSQUIZA, P. S.; HERRERA, J. J. R. Biofilm forming abilityand resistance to industrial disinfectants of Staphylococcus aureus isolated from fishery products. Food Control. v. 39, n. 1, p. 8-16, 2014. Disponível em: <https://doi.org/10.1016/j.foodcont.2013.09.029>. Acesso em: 9 Jun. 2023.
SANTANA, R. F. et al. Qualidade microbiológica de queijo coalho comercializado na cidade de Aracaju, SE. Arquivo Brasileiro de Medicina Veterinária e Zootecnia. v. 60, n. 6, p. 1517-1522, 2008. Disponível em: <https://doi.org/10.1590/S0102-09352008000600031>. Acesso em: 9 Jun. 2023.
SHEPARD, B. D.; GILMORE, M. S. Antibiotic-resistant enterococci: the mechanisms and dynamics of drug introduction and resistance. Microbes and Infection. v. 4, n. 2, p. 215-224, 2002. Disponível em: <10.1016/s1286-4579(01)01530-1>. Acesso em: 9 Jun. 2023.
SHIRTLIFF, M. E.; MADER, J. T.; CAMPER, A. K. Molecular interactions in Biofilms. Chemistry & biology. v. 9, n. 8, p. 859-871, 2002. Disponível em: <https://doi.org/10.1016/S1074-5521(02)00198-9>. Acesso em: 9 Jun. 2023.
SIEVERT, D. M. et al. Antimicrobial-Resistant Pathogens Associated with Healthcare Associated Infections: Summary of Data Reported to the National Healthcare Safety Network at the Centers for Disease Control and Prevention, 2009–2010. Infection Control and Hospital Epidemiology. v. 34, n. 1, p. 1-14, 2013. Disponível em: <10.1086/668770.>. Acesso em: 9 Jun. 2023.
SILVA, A. S.; FURTADO, S. C.; VARGAS, B. L. Avaliação microbiológica do queijo coalho produzido com leite pasteurizado sob refrigeração. Revista Nanbiquara. [s.l.], v. 6, n. 1, p. 118-133, 2017.
SILVA, N.; IGREJAS, G.; GONÇALVES, A.; POETA, P. Commensal gut bacteria: distribution of Enterococcus species and prevalence of Escherichia coli phylogenetic groups in animals and humans in Portugal. Annals of Microbiology. v. 62, pp. 449-459, 2012. Disponível em: <https://doi.org/10.1016/S1074-5521(02)00198-9>. Acesso em: 9 Jun. 2023.
SILVA, R. A. et al. Can artesanal “coalho” cheese from Northeastern Brazil be used as a functional food? Food Chemistry. v. 135, n. 3, pp. 1533-1538, 2012. Disponível em: <10.1016/j.foodchem.2012.06.058>. Acesso em: 9 Jun. 2023.
SOLACHE, M. G.; RICE, L. B. The Enterococcus: a model of Adaptability to Its Environment. Clinical Microbiology Reviews. v. 32, n. 2, 2019. Disponível em: <10.1128/CMR.00058-18>. Acesso em: 9 Jun. 2023.
SOUZA, A. Z. B. et al. Aspectos físico-químicos e microbiológicos do queijo tipo coalho comercializado em estados do nordeste do Brasil. Arquivos do Instituto Biológico. v. 81, n. 1, p. 30-35, 2014. Disponível em: <10.1590/S1808-16572014000100006>. Acesso em: 9 Jun. 2023.
SURYALETHA, K. et al. Decoding the proteomic changes involved in the biofilm formation of Enterococcus faecalis SK460 to elucidate potential biofilm determinants. BMC Microbiology. v. 19, n. 146, 2019. Disponível em: <https://doi.org/10.1186/s12866-019-1527-2>. Acesso em: 9 Jun. 2023.
TEIXEIRA, I. M. S. Biofilmes e infecções associadas a dispositivos médicos. 2019. Dissertação (Mestrado em Ciências Farmacêuticas), Universidade Fernando Pessoa. Portugal.
TEIXEIRA, L.; MERQUIOR, V. L. Molecular typing in bacterial infections. Humana Press. v. 1, n. 12, p. 17-27, 2013. Disponível em: <10.1007/978-1-62703-185-1_2>. Acesso em: 9 Jun. 2023.
TERÁN, C. G.; RODRÍGUEZ, V. R.; RANGEL, A. C. Analysis of antibiotic uses and resistance in an ICU from Monteria, Colombia. Revista médica Risaralda. v. 24, n. 2, pp. 75-80, 2018. Disponível em: <https://dialnet.unirioja.es/servlet/articulo?codigo=6708347>. Acesso em: 9 Jun. 2023.
TERRA, M. R.; FURLANETO, M. C.; MAIA, L. F. Enterococcus multirresistente a antimicrobianos: um importante patógeno nosocomial. Revista Uningá Review. v. 29, n. 3, p. 94- 102, 2017. Disponível em: <https://www.mastereditora.com.br/periodico/20170304_121005.pdf>. Acesso em: 9 Jun. 2023.
TERRA, M. R.; FURLANETO, M. C.; MAIA, L. FOOD AS A POTENTIAL RESERVOIR OF ENTEROCOCCUS THAT HOST DETERMINANTS OF VIRULENCE AND RESISTANCE. Brazilian Journal of Surgery and Clinical Research. v. 22, n. 1, p. 86-93, 2018. Disponível em: <https://www.mastereditora.com.br/periodico/20180303_175405.pdf>. Acesso em: 9 Jun. 2023.
URQUHART, C. et al. Atividade antibiofilme da cumarina frente à Pseudomonas aeruginosa. Anais do 10º Salão Internacional de Ensino, Pesquisa e Extensão, Brazilian Journal of Development. v. 6, n. 10, pp. 77261–77268, 2019. Disponível em: <https://doi.org/10.34117/bjdv6n10-231>. Acesso em: 9 Jun. 2023.
VALLEJO, M.; PARADA, R. B.; MARGUET, E. R. Aislamiento de cepas de Enterococcus hirae productoras de enterocinas a partir del contenido intestinal de mejillón patagônico (Mytilus edulis platensis). Revista Argentina de Microbiologia. v. 52, n. 2, pp. 136-144, 2019. Disponível em: <https://doi.org/10.1016/j.ram.2019.06.001>. Acesso em: 9 Jun. 2023.
VIGNAROLI, C. et al. Multidrug- resistant Enterococci in meat and faeces and co-transfer of resistance from an Enterococcus durans to a human Enterococcus faecium. Current Microbiology. v. 62, n. 5, p. 1438-1447, 2011. Disponível em: <10.1007/s00284-011-9880-x>. Acesso em: 9 Jun. 2023.
WHO – WORLD HEALTH ORGANIZATION. Zoonoses and the Human-Animal-Ecosystems Interface. [S. l.] WHO, 2017.
[1] Doctoranda del Programa de Posgrado en Biociencia Animal. ORCID: 0000-0001-9410-7631. Currículo Lattes: http://lattes.cnpq.br/4532231119888940.
[2] Doctor en Biociencia Animal. ORCID: 0000-0002-2871-6655. Currículo Lattes: http://lattes.cnpq.br/3276511555193502.
[3] Doctor en Biociencia Animal. ORCID: 0000-0002-1982-7142. Currículo Lattes: http://lattes.cnpq.br/5753018560783714.
[4] Licenciada en Medicina Veterinaria. ORCID: 0000-0001-9273-5204. Currículo Lattes: http://lattes.cnpq.br/8329028352662293.
[5] Licenciada en Medicina Veterinaria. ORCID: 0000-0001-5584-9464. Currículo Lattes: http://lattes.cnpq.br/1495170820726310.
[6] Doctoranda del Programa de Posgrado en Biociencia Animal. ORCID: 0000-0003-0698-6125. Currículo Lattes: http://lattes.cnpq.br/5170743479462841.
[7] Doctora en Ciencias Veterinarias. ORCID: 0000-0002-0072-5103. Currículo Lattes: http://lattes.cnpq.br/5676854885081836.
[8] Licenciado en Ciencias Biológicas. ORCID: 0000-0001-7149-4931. Currículo Lattes: https://lattes.cnpq.br/2928291078391850.
[9] M.Sc. en Biociencia Animal. ORCID: 0000-0002-4913-8313. Currículo Lattes: http://lattes.cnpq.br/4967459162060058.
[10] Doctor en Bioquímica y Fisiología, Máster en Fisiología, Biólogo. ORCID: 0000-0003-1493-7964. Currículo Lattes: http://lattes.cnpq.br/9044747136928972.
[11] Orientadora. Doctora del Programa de Posgrado en Biociencia Animal. ORCID: 0000-0002-1289-2902. Currículo Lattes: http://lattes.cnpq.br/5998863169551704.
Enviado: 24 de abril de 2023.
Aprobado: 01 de junio de 2023.