REVUE BIBLIOMETRIQUE
TEDESCHI, Victor Hugo Pancera [1], TSUNODA, Denise Fukumi [2]
TEDESCHI, Victor Hugo Pancera. TSUNODA, Denise Fukumi. Base de données sur la biodiversité : une analyse temporelle de la production scientifique. Revista Científica Multidisciplinar Núcleo do Conhecimento. Année 05, Éd. 09, Vol. 06, p. 68-81. Septembre 2020. ISSN : 2448-0959, Lien d’accès: https://www.nucleodoconhecimento.com.br/biologie/production-scientifique
RÉSUMÉ
La présente recherche porte sur les bases de données biodiversité à travers une enquête bibliographique sur le portail des revues Scopus, utilisant des termes en anglais et des limiteurs dans les mots clés, cherchant à restreindre la recherche à la base de données. Les documents ont été analysés à l’aide de la bibliométrie, dans Biblioshiny (un package R). Un total de 352 documents publiés dans la période de 1984 à 2020 ont été récupérés. Grâce à l’analyse, une augmentation des publications a été observée à partir de l’année 2006. Les chercheurs des États-Unis (54) ont présenté le plus grand nombre de publications, tandis que le Brésil ( 11) est en sixième position. Les résultats trouvés dans cette recherche indiquent une tendance dans les travaux sur le sujet, fournissant ainsi une direction pour les recherches futures.
Mots-clés : Base de données taxonomique, Informatique de la biodiversité, Jeu de données, Bibliométrie.
1. INTRODUCTION
La diversité biologique sur la planète est très élevée et certaines estimations mettent des valeurs en millions. Le Brésil, selon des estimations prudentes, abrite 13 % du biote mondial. Cette estimation est due au fait que le Brésil possède le plus grand système fluvial du monde ainsi que cinq biomes, ce qui a valu au pays le titre de « pays méga diversifié » et compte environ 165 000 espèces connues, et de plus en plus d’espèces sont découvertes chaque jour.
Chaque partie découverte d’une espèce génère des données de manière continue et agrégée sur ses caractéristiques (morphologie, nomenclature, phylogénie, etc.), habitudes (alimentation, comportement, etc.), répartition géographique (observations, registres de collecte, etc.), la génétique (phylogénétique, séquençage de l’ADN, etc.) entre autres.
Toutes ces informations doivent être stockées afin de faciliter leur accès et leur partage dans le monde entier, ce qui justifie la nécessité de développer et de maintenir des bases de données qui effectuent le stockage correct de ces informations ainsi que des systèmes de récupération spécifiques pour la zone. Partant de cette réflexion, l’objectif du présent travail est d’explorer, par une analyse bibliométrique, la production sur le sujet déposée dans le référentiel Scopus qui répertorie les titres académiques à comité de lecture, les titres en libre accès, les actes de congrès et les publications commerciales, parmi autres.
2. FONDEMENT THÉORIQUE
La base de données peut être définie comme un ensemble de données logiquement cohérentes qui a un sens, dont l’interprétation est donnée en fonction de l’application, cet ensemble de données représente abstraitement une partie du monde réel.
L’information biologique peut être divisée en trois dimensions, moléculaire, organisme et écosystème (WILSON, 2005), selon son application cette information est organisée en trois types structuraux de bases, qui sont des bases taxonomiques (organismes), qui se concentrent sur la présentation de la informations morphologiques de l’espèce, bases de l’informatique pour la biodiversité (écosystème), qui a un caractère transdisciplinaire de l’information, afin de fournir des informations écologiques et de distribution géographique, et de la bioinformatique (moléculaire), qui à son tour vise le stockage et la distribution de données moléculaires, de gènes et de protéines.
Les informations qui alimentent ces bases proviennent des processus de numérisation des collections muséales, des rapports de collections, des listes d’enrôlement, extraites des documents publiés, des séquençages des matériaux, etc. Les résultats de cette analyse peuvent être stockés ou partagés dans une variété de formats tels que des documents texte, des feuilles de calcul, des pages Web, des bases de données connexes, des cartes ou SIG (Système d’Information Géographique), des images, etc. (FRAZIER; WALL; GRANT, 2008)
La numérisation de ces matériaux est d’une importance vitale pour la préservation et le partage des informations sur les espèces, mais cette approche apporte de nouveaux défis, tels que la fiabilité des données numérisées, à partir de ce point, Ruas (2017) souligne l’importance des métadonnées, qui sont informations qui Celles-ci décrivent les informations contenues dans la base de données, dans la conservation du contenu généré par les processus de numérisation, les méta-informations garantissent la fidélité et l’authenticité des informations présentées, ainsi que la contextualisation et que les informations ont été capturées.
3. COURS MÉTHODOLOGIQUE
Le choix des termes à utiliser dans cette recherche a été effectué en expérimentant différents ensembles de mots booléens et d’opérateurs, basés sur Scopus. Le flux de recherche (du niveau général au niveau spécifique) et le nombre de documents récupérés dans chaque expérience sont présentés dans le tableau 1.
Tableau 1 – Ensembles de mots et opérateurs booléens utilisés dans la base de données Scopus et nombre de documents récupérés
| Mots et opérateurs | Documents récupérés |
| ALL ( biodiversity AND database ) | 91.714 |
| ALL ( taxonomy AND database ) | 113.470 |
| ALL ( biodiversity OR taxonomy AND database OR dataset ) | 226.573 |
| ALL ( “biodiversity database” ) | 1.487 |
| ALL ( “biodiversity database” OR “taxonomy database” ) | 2.158 |
| ALL ( “biodiversity database” OR “taxonomy database” ) AND ( LIMIT-TO ( EXACTKEYWORD , “Biodiversity” ) OR LIMIT-TO ( EXACTKEYWORD , “Taxonomy” ) OR LIMIT-TO ( EXACTKEYWORD , “Databases, Genetic” ) OR LIMIT-TO ( EXACTKEYWORD , “Factual Database” ) OR LIMIT-TO ( EXACTKEYWORD , “Data Set” ) OR LIMIT-TO ( EXACTKEYWORD , “Protein Database” ) OR LIMIT-TO ( EXACTKEYWORD , “Databases, Protein” ) OR LIMIT-TO ( EXACTKEYWORD , “Biodiversity Informatics” ) OR LIMIT-TO ( EXACTKEYWORD , “Data Quality” ) ) AND ( EXCLUDE ( SUBJAREA , “IMMU” ) OR EXCLUDE ( SUBJAREA , “MEDI” ) OR EXCLUDE ( SUBJAREA , “NEUR” ) OR EXCLUDE ( SUBJAREA , “PHYS” ) OR EXCLUDE ( SUBJAREA , “PHAR” ) OR EXCLUDE ( SUBJAREA , “ARTS” ) OR EXCLUDE ( SUBJAREA , “VETE” ) OR EXCLUDE ( SUBJAREA , “HEAL” ) OR EXCLUDE ( SUBJAREA , “NURS” ) ) | 723 |
Source : Préparé par les auteurs (2020).
A partir des résultats trouvés, les 100 premiers documents ont été lus, afin d’observer si les résultats étaient alignés avec le thème abordé dans la présente recherche, apportant ainsi des ajustements aux termes afin d’affiner les résultats. Comme on peut le voir dans le tableau 1, dans certaines combinaisons de termes, le nombre de documents récupérés variait de 700 à plus de 200 000 documents. Cette étape était nécessaire car des termes tels que biodiversity et taxonomic sont utilisés dans des contextes différents par les chercheurs en sciences biologiques.
La stratégie de recherche dans la base de données Scopus (Elsevier) était la recherche des termes anglais « biodiversity database » et « taxonomic database », dans tous les champs, un limiteur a été utilisé dans les mots clés, en sélectionnant ceux qui sont liés aux données, les mots sélectionnés sont « Database », « Data set », « Data Base », « database, factual », « factual database », « Database Systems », « data quality » et « data management», en utilisant les résultats obtenus à ce jour à partir de la recherche du 28 juin 2020. Les paramètres de requête avec les codes de champ et les opérateurs ont donné :
L’analyse métrique de la fréquence des mots (Titre, résumé, mots-clés des auteurs, mots-clés étendus), de la fréquence de production des documents (pays, sources, auteurs) et de l’évolution dans le temps (Production par année, utilisation des mots-clés) a été réalisée à l’aide de Biblioshiny (interface graphique du package Bibliometrix, réalisé en langage R) et l’éditeur de tableur Microsoft Excel. Les données ont été exportées directement de la base de données Scopus au format CSV, compatible avec la bibliothèque Biblioshiny pour le logiciel R v.3.6.3 et Microsoft Excel 2016.
4. PRÉSENTATION ET DISCUSSION DES RÉSULTATS
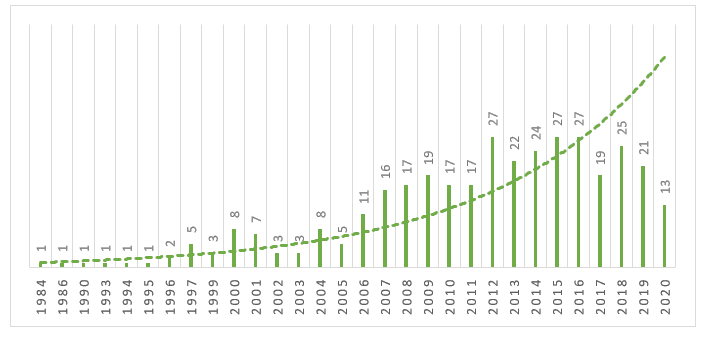
L’analyse comprend les 352 documents récupérés lors de la recherche dans la base de données Scopus le 28 juin 2020, dont 272 articles publiés, 30 actes de conférence et un chapitre de livre. La première publication trouvée fait référence à l’année 1984. Les 352 documents récupérés ont été écrits par 1 978 auteurs. 2 068 mots-clés étendus (mots générés automatiquement par la base de données) et 1 033 mots-clés d’auteurs ont été identifiés.
Comme le montre le graphique 1, on observe que dans l’intervalle de temps entre 1984 et 1996, il n’y a pas eu de production significative sur le sujet, après cette période on observe une augmentation du nombre de productions sur le sujet. On observe qu’en 2017 il y a eu une oscillation importante, où il y a eu une baisse de la production, et une reprise l’année suivante. Jusqu’au moment de cette recherche 2020 présente déjà 13 articles publiés.
Graphique 1 – Evolution de la production scientifique dans la base Scopus de 1984 à 2020
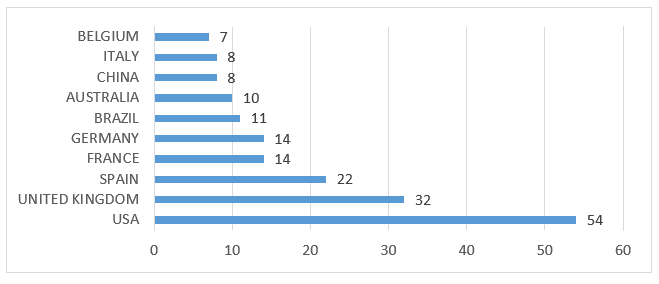
La répartition des documents par pays fait apparaître les États-Unis d’Amérique (54) avec le plus grand nombre de publications dans ce domaine, suivis du Royaume-Uni (32), qui affiche une prédominance de documents en langue anglaise. Ce résultat peut avoir été influencé par l’utilisation de termes de recherche en anglais. En revanche, plusieurs revues dans différentes langues utilisent le abstract comme l’un des éléments obligatoires. Le Brésil (11) apparaît à la sixième place en nombre de publications, comme le montre le graphique 2.
Graphique 2 – Contribution de la production scientifique dans le monde
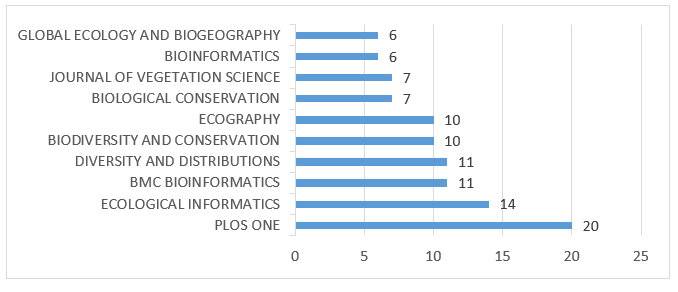
Parmi les 352 documents récupérés dans la requête, PLOS ONE a présenté le plus grand nombre d’articles publiés, sur un total de 20, suivi par Ecological Informatics avec 14. Le graphique 3 présente les 10 sources avec le plus grand nombre de publications.
Graphique 3 – Sources avec le plus grand nombre de productions
Les 10 chercheurs les plus productifs sont présentés dans le tableau 2 et les 10 les plus cités dans le tableau 3. L’auteur le plus productif (10) et le plus cité (241) est le Dr Jorge M. Lobo, professeur de recherche au Département de Biogeography and Global Change do Museo Nacional de Ciencias Naturales, à Madrid, Espagne. L’article le plus référencé de l’auteur est « Use of niche models in invasive species risk assessments » co-écrit avec les chercheurs A. Jiménez-Valverde, A. T. Peterson, J. Soberón, J. M. Overton et P. Argón, qui est également le troisième article le plus cité. (384), présenté dans le tableau 4. Ce travail analyse l’utilisation des données de localisation des espèces pour l’élaboration de modèles prédictifs du risque d’installation d’espèces envahissantes courantes dans les grands ensembles de données sur la biodiversité, telles que les données de géoréférencement et les erreurs d’identification des espèces.
Tableau 2 – Auteurs les plus productifs
| Autores | Artigos |
| LOBO JM | 10 |
| HORTAL J | 9 |
| PAGE RDM | 7 |
| SOBERóN J | 6 |
| BOOTH TH | 5 |
| COSTELLO MJ | 5 |
| KREFT H | 5 |
| ARIñO AH | 4 |
| GURALNICK R | 4 |
Source : Préparé par les auteurs (2020).
Tableau 3 – Auteurs les plus cités
| Autores | Citações |
| LOBO J M | 241 |
| PETERSON A T | 223 |
| HORTAL J | 217 |
| GUISAN A | 132 |
| COSTELLO M J | 117 |
| GRAHAM C H | 114 |
| SOBERÃ N J | 109 |
| FERRIER S | 108 |
| JIMÃ NEZ VALVERDE A | 108 |
Source : Préparé par les auteurs (2020).
Les cinq articles les plus référencés sont listés dans le tableau 4. Le plus cité est l’article intitulé « SequenceMatrix: concatenation software for the fast assembly of multi-gene datasets with character set and codon information », produit par Vaidya, G., Lohman, D. J. et Meier, R. publié en 2011. Dans ce travail, les auteurs présentent le logiciel SequenceMatrix utilisé dans l’analyse et l’association de plusieurs gènes provenant de différents jeux de données, en soulignant la facilité d’utilisation comme point fort et en présentant ses principales fonctionnalités. Le logiciel permet des fonctionnalités de détection et de correction des erreurs contenues dans les jeux de données.
Andelman et Fagan (2000) évaluent si l’utilisation d’espèces appelées « drapeau » ou « parapluie » sont efficaces dans l’utilisation comme substituts dans la conservation, car au lieu de se concentrer sur la conservation de plusieurs zones, l’accent est mis sur la conservation de ces zones peu d’espèces, ce qui contribue par conséquent à la conversation de zones entières. Pour tester leur hypothèse, les auteurs ont utilisé trois bases de données avec différentes dimensions de couverture.
Jayasiri et al. (2015) traitent dans leur article de la création d’une base de données via le web, centrée sur la diversité des champignons, afin d’améliorer la précision des noms scientifiques, en mettant l’accent sur la taxonomie. La base compte 76 conservateurs spécialisés dans les différents groupes, assurant ainsi la fiabilité des données.
Lobo, Jiménez-Valverde et Hortal (2010) traitent des données d’absence d’espèces dans certaines régions contenues dans les bases de données, dans la génération de modèles de distribution. Ils ont réalisé une étude de cas d’une espèce de coléoptère dont la distribution est connue, afin de démontrer les erreurs possibles dans l’utilisation de ces données d’absence et leur importance dans la modélisation des cartes de distribution.
Tableau 4 – les cinq articles les plus cités
| Des articles | Total de citations |
| VAIDYA, G.; LOHMAN, D. J.; MEIER, R. SequenceMatrix: Concatenation software for the fast assembly of multi-gene datasets with character set and codon information. Cladistics, v. 27, n. 2, p. 171–180, 2011. | 847 |
| ANDELMAN, S. J.; FAGAN, W. F. Umbrellas and flagships: Efficient conservation surrogates or expensive mistakes? Proceedings of the National Academy of Sciences of the United States of America, v. 97, n. 11, p. 5954–5959, 2000. | 447 |
| JIMÉNEZ-VALVERDE, A. et al. Use of niche models in invasive species risk assessments. Biological Invasions, v. 13, n. 12, p. 2785–2797, 2011. | 384 |
| JAYASIRI, S. C. et al. The Faces of Fungi database: fungal names linked with morphology, phylogeny and human impacts. Fungal Diversity, v. 74, n. 1, p. 3–18, 2015. | 355 |
| LOBO, J. M.; JIMÉNEZ-VALVERDE, A.; HORTAL, J. The uncertain nature of absences and their importance in species distribution modelling. Ecography, v. 33, n. 1, p. 103–114, 2010. | 325 |
Source : Préparé par les auteurs (2020).
Les cinq articles les plus cités dans les documents sont présentés dans le tableau 5. L’article le plus cité dans les documents est « Interoperability of biodiversity databases: biodiversity information on every desktop », produit par les auteurs Edwards, Lane et Nielsen et publié dans l’année 2000, dans l’article et le traité sur le GBIF, qui a été créé pour faciliter la numérisation des données sur la biodiversité, et les rendre librement accessibles. Dans l’article est présenté le GBIF et les perspectives futures des données sur la biodiversité.
Hortal, Lobo et Jiménez-Valverde (2007) présentent une étude de cas sur les limites rencontrées dans les bases de données sur la biodiversité, en se concentrant sur la diversité des semences et des plantes. Dans leur travail Soberón et Peterson (2004), ils discutent du potentiel de l’informatique de la biodiversité, dans leur discussion, ils commentent l’application des méthodes de gestion de la biodiversité, et pas seulement pour les études fondamentales et le partage d’informations. Soberón et al. (2006), dans leur article, démontrent l’utilisation des données sur la biodiversité présentes dans les bases pour estimer la richesse et dans différentes résolutions de répartition géographique. Bisby (2000) traite de l’émergence de grands systèmes mondiaux d’information biologique dans ses recherches.
Tableau 5 – les 5 documents les plus cités dans les références de la période
| Documents cités dans les références | Nombre de citations |
| EDWARDS, James L.; LANE, Meredith A.; NIELSEN, Ebbe S. Interoperability of biodiversity databases: biodiversity information on every desktop. Science, v. 289, n. 5488, p. 2312-2314, 2000. | 49 |
| HORTAL, Joaquín; LOBO, Jorge M.; JIMÉNEZ‐VALVERDE, ALBERTO. Limitations of biodiversity databases: case study on seed‐plant diversity in Tenerife, Canary Islands. Conservation Biology, v. 21, n. 3, p. 853-863, 2007. | 37 |
| SOBERÓN, Jorge; PETERSON, Townsend. Biodiversity informatics: managing and applying primary biodiversity data. Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences, v. 359, n. 1444, p. 689-698, 2004. | 34 |
| SOBERÓN, Jorge et al. Assessing completeness of biodiversity databases at different spatial scales. Ecography, v. 30, n. 1, p. 152-160, 2007. | 25 |
| BISBY, Frank A. The quiet revolution: biodiversity informatics and the internet. Science, v. 289, n. 5488, p. 2309-2312, 2000. | 23 |
Source : Préparé par les auteurs (2020).
Les mots-clés les plus utilisés par les auteurs Database (49), suivis de la biodiversity (32), de la taxonomy (26), de l’biodiversity informatics (19) et de la data quality (19), ces fréquences de mots ont probablement été affectées par la méthodologie de sélection des documents effectuée dans cette recherche. La figure 1 montre les 50 mots-clés avec l’occurrence la plus élevée du 1033.
Figure 1 – Les 50 mots-clés les plus fréquents de l’auteur

Concernant les mots clés étendus, le mot le plus utilisé est Biodiversity (236), suivi de database (125), Taxonomy (88), dataset (66) et classification (62). Et dans les résumés les mots les plus fréquents data (1 221), species (856), biodiversity (488), database (315), databases (268). Les mots les plus fréquents dans les titres des documents sont data (113), biodiversity (100), species (68), database (57), databases (44).
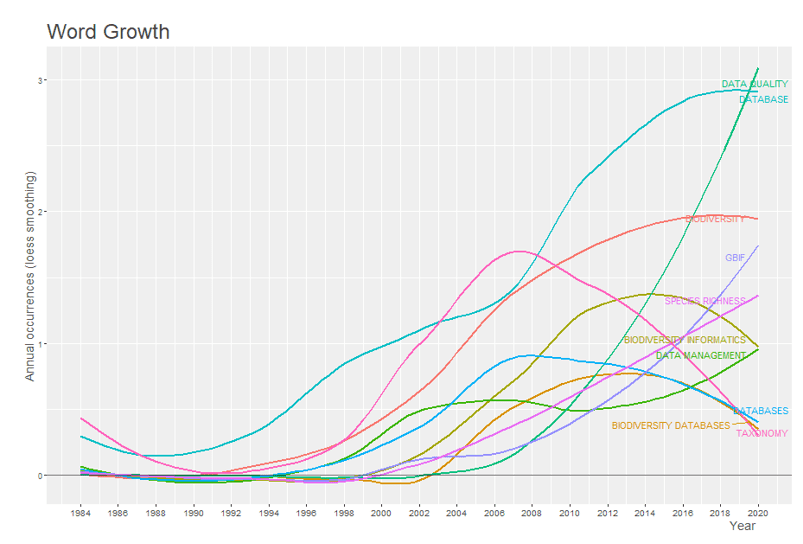
Les 10 mots-clés des auteurs avec les fréquences les plus élevées sont représentés en fonction du nombre d’occurrences accumulées au fil du temps dans le graphique 4. Dans le graphique, il est également possible d’observer la croissance exponentielle du mot data quality, ce qui peut indiquer une augmentation de l’intérêt par des études visant à évaluer les informations contenues dans les bases de données existantes, ainsi que ce mot et à observer la croissance du mot GBIF, qui correspond au Global Biodiversity Information Facility, qui est un réseau international de partage de données sur tous les types de vie dans la terre. Les mots database, biodiversity database, databases et biodiversity informatics ont connu une baisse d’utilisation ces dernières années.
Graphique 4 – Dynamique des mots clés de l’auteur dans le temps
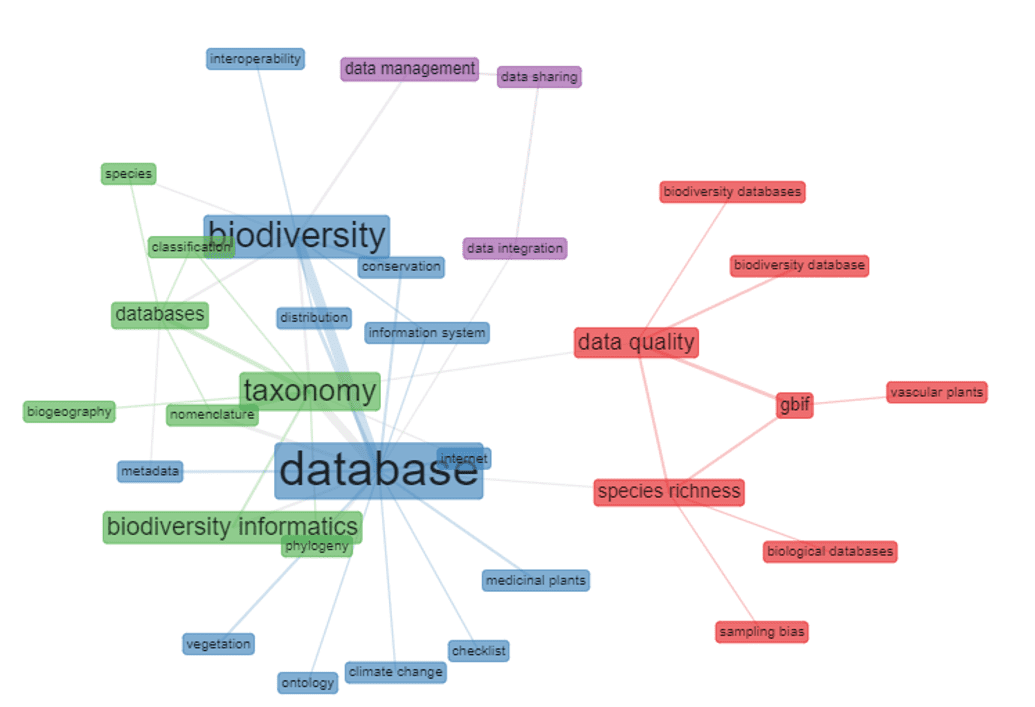
Dans l’analyse des cooccurrences des mots-clés, certaines relations peuvent être perçues : a) database – biodiversity – metadata, internet, information system, distribution, conservation et checklist ; b) taxonomy – biodiversity informatics et phylogeny – databases – species, classification, nomenclature et biogeography ; c) data quality – species richness – biodiversity databases, biodiversity database, GBIF et biological databases ; d) data management, datashring et data integration. Il convient de souligner les relations présentées dans l’ensemble « c », dans lequel il est possible d’observer l’accent mis sur la qualité des données présentées dans les bases, avec un accent sur le GBIF. La séparation entre l’ensemble « a » et « b » se présente de manière intéressante, rendant visible la différence dans l’objet de la recherche, où l’ensemble « a » se concentrait sur la distribution, la conservation et la diversité des espèces, tandis que le l’ensemble « b » traite davantage de la nomenclature et de la classification des espèces. La figure 2 présente les relations susmentionnées.
Figure 2 – La cooccurrence des mots-clés utilisés par les auteurs
5. CONSIDÉRATIONS PARTIELLES
La présente étude a présenté l’évolution des productions scientifiques liées aux bases de données de biodiversité et de taxonomie présentes dans la base de données Scopus. La requête a renvoyé 352 documents, distribués dans la période de 1984 à 2020. Il y a eu une augmentation des publications à partir de 2006.
Selon les informations obtenues grâce aux analyses, il a été possible de visualiser la croissance des productions et les pays qui ont le plus produit sur le sujet, et a permis de pointer les publications les plus citées référencées.
L’identification des mots-clés les plus utilisés a peut-être été affectée par la méthodologie appliquée dans cette recherche, puisqu’ils ont été utilisés dans les termes de recherche appliqués dans la base de données Scopus. Cependant, il est possible d’observer une croissance de l’intérêt pour les documents récupérés liés à la qualité des données trouvées dans ces bases.
Cet article présente une première approche exploratoire des bases de données liées à la biodiversité. Sur la base des résultats, les questions liées à la qualité des données présentes dans les bases de données les plus utilisées, telles que la base de données GBIF, seront mieux explorées à l’avenir. Un autre point à aborder dans les recherches futures est la forme de représentation des espèces brésiliennes dans ces bases de données et la possibilité de récupérer ces informations par les parties intéressées.
La continuité de la recherche est conforme aux intérêts proposés dans la thèse de l’auteur, dans laquelle il propose le développement d’une base de données/portail en libre accès, qui facilite la diffusion de données sur la diversité de la faune brésilienne (animaux), valorisant des données de qualité en constante mise à jour et confiance, pour répondre aux besoins des professionnels et des chercheurs dans les domaines biologiques.
RÉFÉRENCES
ANDELMAN, S. J.; FAGAN, W. F. Umbrellas and flagships: Efficient conservation surrogates or expensive mistakes? Proceedings of the National Academy of Sciences of the United States of America, v. 97, n. 11, p. 5954–5959, 2000.
BISBY, Frank A. The quiet revolution: biodiversity informatics and the internet. Science, v. 289, n. 5488, p. 2309-2312, 2000.
EDWARDS, James L.; LANE, Meredith A.; NIELSEN, Ebbe S. Interoperability of biodiversity databases: biodiversity information on every desktop. Science, v. 289, n. 5488, p. 2312-2314, 2000.
FRAZIER, C.K., WALL, J.; GRANT, S.. Initiating a Natural History CollectionDigitisation Project, version 1.0. Copenhagen: Global Biodiversity Information Facility.75 pp. 2008.
HORTAL, Joaquín; LOBO, Jorge M.; JIMÉNEZ‐VALVERDE, ALBERTO. Limitations of biodiversity databases: case study on seed‐plant diversity in Tenerife, Canary Islands. Conservation Biology, v. 21, n. 3, p. 853-863, 2007.
JAYASIRI, S. C. et al. The Faces of Fungi database: fungal names linked with morphology, phylogeny and human impacts. Fungal Diversity, v. 74, n. 1, p. 3–18, 2015.
JIMÉNEZ-VALVERDE, Alberto et al. Use of niche models in invasive species risk assessments. Biological invasions, v. 13, n. 12, p. 2785-2797, 2011.
LOBO, J. M.; JIMÉNEZ-VALVERDE, A.; HORTAL, J. The uncertain nature of absences and their importance in species distribution modelling. Ecography, v. 33, n. 1, p. 103–114, 2010.
RUA, J. DIGITALIZAÇÃO, PRESERVAÇÃO E ACESSO: contributos para o projeto Museu Digital da U.PORTO. Páginas a&b. S.3, nº especial (2017) 199-229 | DOI 10.21747/21836671/pag2017a13
SOBERÓN, Jorge et al. Assessing completeness of biodiversity databases at different spatial scales. Ecography, v. 30, n. 1, p. 152-160, 2007.
SOBERÓN, Jorge; PETERSON, Townsend. Biodiversity informatics: managing and applying primary biodiversity data. Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences, v. 359, n. 1444, p. 689-698, 2004.
VAIDYA, G.; LOHMAN, D. J.; MEIER, R. SequenceMatrix: Concatenation software for the fast assembly of multi-gene datasets with character set and codon information. Cladistics, v. 27, n. 2, p. 171–180, 2011.
WILSON, Edward O. Systematics and the future of biology. Proceedings of the National Academy of Sciences, v. 102, n. suppl 1, p. 6520-6521, 2005.
[1] Diplômée en Sciences Biologiques, Pontifícia Universidade Católica do Paraná (PUC-PR), Paraná, Brésil. Diplômé en électronique industrielle, Faculdade de Tecnologia de Curitiba (FATEC) Curitiba, Paraná, Brésil. Étudiant en Master en Gestion de l’Information, Universidade Federal do Paraná (UFPR), Curitiba, Paraná, Brésil.
[2] Doctorat en génie électrique et informatique, Universidade Tecnológica Federal do Paraná (UTFPR), Curitiba, Paraná, Brésil.
Envoyé : Septembre 2020.
Approuvé : Septembre 2020.